Ringkasan
FD, faktor transkripsi bZIP yang dilestarikan, terkenal karena perannya dalam mendorong pembungaan. Di sini, kami mengungkap bahwa urutan pengkodean pendek dari gen FD Chrysanthemum morifolium , CmFDa , menekan pembungaan. Menariknya, kami mengidentifikasi CmFDa sebagai epialel baru yang diatur secara ketat oleh metilasi DNA pada krisan untuk pertama kalinya. Pengeditan epigenetik promotor CmFDa menggunakan CRISPR-dCas9-TET1cd untuk demetilasi atau dCas9-SunTag-NtDRM2cd untuk metilasi masing-masing dapat menekan atau mendorong pembungaan. CmFDa secara epigenetik menekan transisi bunga dengan menekan ekspresi aktivator bunga CmSOC1 dan CmAP1 . Kami menunjukkan bahwa CmFDa merekrut kompleks represif Polycomb 2 (PRC2) ke promotor CmSOC1 dan CmAP1 . Hasil ini menunjukkan bahwa CmFDa merupakan penghambat pembungaan melalui represi epigenetik gen integrator pembungaan. Studi ini memberikan strategi baru untuk perbaikan epigenetik waktu pembungaan pada tanaman.
Perkenalan
Krisan ( Chrysanthemum morifolium ) adalah salah satu bunga yang paling bernilai di seluruh dunia dan memegang posisi penting dalam industri hortikultura. Mengatur periode mekar untuk memenuhi tujuan produksi tahunan yang ditargetkan sangat penting untuk tanaman bunga potong seperti krisan (Su et al ., 2019 ). Oleh karena itu, waktu berbunga adalah salah satu target utama pemuliaan krisan. Faktor biologis dan non-biologis utama yang mempengaruhi waktu berbunga meliputi fotoperiode, suhu lingkungan, hormon tanaman, pasokan nutrisi dan jalur pembungaan otonom. Interaksi antara faktor-faktor ini mengatur ekspresi integrator jalur pembungaan, seperti kompleks FT-FD dan SOC1 (Takagi et al ., 2023 ). FD, anggota keluarga protein bZIP, memainkan peran penting dalam modulasi transisi bunga pada angiospermae. Melalui domain pengikat DNA-nya, FD, bersama dengan faktor transkripsi lainnya, mengikat promotor gen target untuk mengatur tingkat transkripsinya. Banyak efek awal FD pada ekspresi gen di meristem apikal pucuk (SAM) dan primordia bunga merupakan hasil pengikatan langsungnya ke promotor gen target (misalnya, FT , TFL1 , AP1 , SOC1 , dan FUL ) dan perekrutan faktor terkait pembungaan berikutnya (Abe et al ., 2005 ; Collani et al ., 2019 ; Yue et al ., 2021 ; Zhu et al ., 2020 ). Mesin molekuler yang rumit ini menggarisbawahi sifat kompleks dari regulasi pembungaan, dengan mekanisme yang tepat masih sulit dipahami.
Investigasi ekstensif mekanisme pembungaan tanaman dalam beberapa tahun terakhir telah mengungkapkan bahwa faktor epigenetik, seperti metilasi DNA dan modifikasi histon, memainkan peran penting dalam memodulasi ekspresi gen pembungaan utama (He et al ., 2020 ; Shi et al ., 2023 ). Studi terbaru mengungkapkan bahwa beragam faktor transkripsi melibatkan berbagai pengubah kromatin untuk mengatur beberapa gen pengatur pembungaan utama termasuk FLC dan FT sebagai respons terhadap isyarat musiman. PRC2 terutama memengaruhi proses pembungaan dengan mengatur ekspresi gen penekan pembungaan FLC melalui jalur yang bergantung pada suhu, di mana komponen inti PRC2, termasuk CLF dan SWN (metiltransferase H3K27me3), VRN2, FIE dan MSI1, merespons vernalisasi untuk membungkam ekspresi FLC (Buzas et al ., 2011 ; Sung dan Amasino, 2004 ; Zhang et al ., 2020 ). CLF, LHP1 dan EMF1 mengatur pembungkaman transkripsi gen FT (Li et al ., 2018 ; Wang et al ., 2014 ), di mana CLF direkrut ke daerah promoter gen FT oleh faktor transkripsi NF-Y (Cao et al ., 2014 ; Luo et al ., 2018 ). Faktor transkripsi bertindak sebagai jembatan yang menghubungkan berbagai faktor pengatur pembungaan, dan memahami aksi sinergis faktor transkripsi dan protein modifikasi kromatin PRC2 sangat penting untuk mengatur pembungaan. Lebih jauh, mutan met1/drm1/drm2/cmt3/cmt2 (mddcc) di Arabidopsis tidak dapat berbunga dalam kondisi hari panjang (LD), yang menunjukkan peran penting metilasi DNA dalam pengaturan pembungaan (He et al ., 2022 ). FWA adalah salah satu gen yang paling banyak dipelajari yang menghubungkan metilasi DNA dengan pengaturan pencetakan. Hilangnya metilasi sitosin (5 m C) pada promotor FWA menghasilkan epialel fwa dominan dengan ekspresi yang meningkat dan fenotipe pembungaan akhir yang dapat diwariskan (Zhang et al ., 2018 ).
Epigenetika adalah studi tentang perubahan yang diwariskan dalam ekspresi gen yang disebabkan bukan oleh variasi urutan genom tetapi oleh metilasi DNA dan modifikasi histon (Baulcombe dan Dean, 2014 ; Cavalli dan Heard, 2019 ; Springer dan Schmitz, 2017 ). Pemuliaan epigenetik, sebuah metode yang sedang berkembang, memungkinkan produksi sifat-sifat yang diwariskan pada tanaman poliploid melalui reproduksi aseksual. Ketika jenis modifikasi epigenetik dari gen target diidentifikasi, pemuliaan epigenetik dapat lebih menyempurnakan fenotipe tanaman melalui pengeditan epigenom yang tepat. Pada krisan, Cas9 yang dinonaktifkan (dCas9) yang menyatu dengan TET1cd baru-baru ini digunakan untuk memberantas metilasi DNA dari daerah promotor epialel CmMYB6 , yang memengaruhi warna bunga, sehingga mengubah rona bunga (Tang et al ., 2022 ). Hal ini mengarahkan kami untuk meneliti apakah epimutan stabil dapat dihasilkan pada tanaman menggunakan penghambat epigenetik dan apakah fenotipe tanaman dapat ditingkatkan menggunakan teknik pemuliaan epigenetik.
Dalam penelitian ini, 5-azacytidine (5-AzaC), zat kimia yang banyak digunakan untuk menginduksi perubahan tingkat metilasi DNA global pada organisme yang luas, digunakan untuk memutagenkan tanaman krisan. Akibatnya, mutan dengan pembungaan tertunda diidentifikasi dan dipelajari. Epialel yang bertanggung jawab untuk berbagai fenotipe pembungaan pada tanaman yang sama diidentifikasi sebagai CmFDa untuk pertama kalinya. Tingkat metilasi DNA pada promotor CmFDa memengaruhi ekspresi gen dan waktu pembungaan, dan kedua fenotipe ini dapat diwariskan secara mitosis. Penelitian ini menjelaskan peran epigenetik dan genetik CmFDa dalam inisiasi pembungaan pada krisan. Selain itu, alat penyunting berbasis CRISPR/Cas9 yang mengandung dCas9 yang menyatu dengan domain katalitik (CD) dari metiltransferase DRM2 (DRM2cd) atau demetilase TET1 (TET1cd) digunakan untuk memvalidasi waktu pembungaan yang berubah, yang menjadi dasar teknis untuk peningkatan waktu pembungaan pada krisan melalui modifikasi epigenetik. Studi ini juga mengungkap bahwa CmFDa berinteraksi dengan PRC2 untuk memediasi pengendapan H3K27me3 pada promotor gen pengaktif pembungaan CmSOC1a dan CmAP1a , yang menyebabkan ekspresi gen yang ditekan dan pembungaan yang tertunda, sehingga membangun dasar teoritis untuk pemuliaan epigenetik tanaman dengan waktu pembungaan yang lebih baik.
Hasil
Epimutan krisan yang dihasilkan menggunakan 5-AzaC menunjukkan fenotipe pembungaan tertunda yang dapat diwariskan
Untuk menyusun pustaka mutan epigenetik yang lengkap, kami memperlakukan total 1650 tanaman krisan yang termasuk dalam 10 varietas berbeda dengan 5-AzaC. Sebanyak 805 tanaman yang diperlakukan diperoleh, yang menunjukkan variasi fenotipik dalam sifat agronomi utama termasuk perkembangan (Gambar 1a ), bentuk daun (Gambar 1b ), warna daun (Gambar 1c ), warna bunga (Gambar 1d ) dan waktu pembungaan (Gambar 1e,f ).

Kami memilih mutan dengan waktu pembungaan yang diubah untuk penelitian lebih lanjut (Gambar 1f ). Dibandingkan dengan tipe liar (WT), mutan yang diinduksi 5-AzaC menunjukkan fenotipe pembungaan yang tertunda dan jumlah daun yang secara signifikan lebih tinggi (Gambar 1g ). Melalui perbanyakan vegetatif kuncup akar (Gambar 1h ), kami memperoleh dua tanaman: satu yang berbunga pada saat yang sama dengan WT (dinamai sebagai galur WT) dan yang lain yang menunjukkan pembungaan yang tertunda (dinamai sebagai galur epilat ) (Gambar 1i ). Galur WT membentuk kuncup bunga pada 100–125 hari setelah tanam, sedangkan galur epilat tidak (Gambar 1j ). Selain itu, galur epilat menunjukkan jumlah daun yang secara signifikan lebih tinggi daripada galur WT (Gambar 1k ). Uji lapangan lebih lanjut mengonfirmasi stabilitas fenotipe pembungaan di seluruh tanaman epimutan yang dihasilkan dari epilat (Gambar 1l,m ). Pengamatan berkelanjutan terhadap keturunan galur WT dan galur epilat pada generasi kedua (Gambar 1n ) dan generasi ketiga (Gambar 1o ) menunjukkan bahwa perbedaan fenotip waktu pembungaan antara kedua genotip tersebut stabil. Hal ini membuktikan bahwa fenotip mutan yang diperoleh dengan menggunakan 5-AzaC bersifat heritabel.
Untuk membuktikan bahwa galur WT dan epilate memiliki asal yang sama dan berbagi latar belakang genetik yang sama, kami melakukan enam galur krisan yang dipilih secara acak (‘ZT’, ‘BY’, ‘FSD’, ‘DXYP’, ‘FD’ dan ‘YX’) serta galur WT dan epilate pada analisis sekuensing DNA terkait situs restriksi (RAD-seq). Hasilnya menunjukkan bahwa galur WT dan epilate mengelompok bersama, sementara enam sampel lainnya tersebar dan berjauhan dari galur WT dan epilate (Gambar 1p ). Verifikasi berbasis penanda sekuens polimorfik amplifikasi terbelah (CAPS) dari dua fragmen genom yang diperoleh secara acak melalui analisis SNP genom utuh tidak mengungkapkan perbedaan signifikan antara galur WT dan epilate tetapi membedakan kedua galur ini dari enam galur lainnya (Gambar 1q ). Hasil ini menunjukkan bahwa galur WT dan epilate memiliki latar belakang genetik yang sama, mengesampingkan kemungkinan bahwa perbedaan waktu pembungaannya disebabkan oleh mutasi genetik.
Garis mutan epilat menunjukkan penurunan tingkat metilasi DNA global
Bahasa Indonesia: Untuk lebih jauh mengonfirmasi efektivitas mutasi epigenetik yang diinduksi oleh 5-AzaC, kami memeriksa tingkat metilasi DNA di seluruh genom pada garis WT dan epilate . Sebanyak 257 Gb pembacaan bersih diperoleh. Berikutnya, pembacaan bersih diselaraskan dengan urutan genom krisan yang dibudidayakan ‘Zhongshanzigui’, dan metilasi sitosin dideteksi menggunakan perangkat lunak bismark (Tabel S1 ). Peta resolusi basa tunggal krisan menunjukkan tingkat metilasi DNA di setiap kromosom serta kepadatan gen dan transposon di situs yang sesuai. Kepadatan gen umumnya berkorelasi negatif dengan tingkat metilasi DNA dalam konteks CG, CHG dan CHH (Gambar 2a ), dan tingkat metilasi CHH secara signifikan lebih rendah pada epilate daripada pada garis WT (Gambar 2a ). Secara keseluruhan, tingkat metilasi sitosin pada garis epilat lebih rendah dibandingkan dengan garis WT (Gambar 2a ; Tabel S1 ).
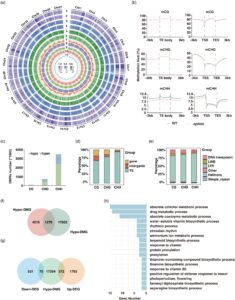
Berikutnya, kami menganalisis perbedaan tingkat metilasi pada gen dan daerah elemen transposabel (TE) antara garis WT dan epilat (Gambar 2b ). Hasilnya menunjukkan bahwa tingkat metilasi CHH di daerah badan gen, TE, dan daerah pengapitnya secara signifikan lebih rendah dalam sampel epilat daripada dalam sampel WT (Gambar 2b ). Selain itu, dibandingkan dengan WT dan terhadap latar belakang ketiga konteks sitosin, jumlah daerah hipometilasi lebih besar daripada daerah hipermetilasi dalam sampel epilat . CHH-hipo-DMR adalah tipe DMR yang paling melimpah (Gambar 2c ). Analisis distribusi genom hipo-DMR menunjukkan bahwa sebagian besar DMR ini, terutama 88% DMR tipe CHH, terletak di daerah TE (Gambar 2d ). Selain itu, di antara berbagai jenis TE, transposon tipe LTR mengandung mayoritas hipo-DMR (Gambar 2e ).
Untuk menggambarkan gen yang diatur oleh DMR dengan lebih baik, kami mendefinisikan gen dengan DMR di daerah promoter (0 hingga -3000 bp) sebagai gen metilasi diferensial (DMG). Sebanyak 19.081 gen dikaitkan dengan DMR tipe hipo, sementara 5.794 gen dikaitkan dengan DMR tipe hiper (Gambar 2f ). Di antara kedua set gen ini, 1.279 gen diatur oleh DMR hiper dan hipo. Gen yang dipengaruhi semata-mata oleh kadar metilasi yang meningkat pada epilat (4.515) ditetapkan sebagai Hiper-DMG, dan gen yang dipengaruhi secara eksklusif oleh kadar metilasi yang menurun pada epilat (17.802) ditetapkan sebagai Hipo-DMG (Gambar 2f ). Ketimpangan dalam jumlah Hyper-DMG dan Hypo-DMG ini menunjukkan bahwa ciri fenotipik garis epilat mungkin terkait dengan pengurangan kadar metilasi globalnya dibandingkan dengan garis WT.
Berikutnya, kami menggunakan diagram Venn untuk menganalisis hubungan antara gen dengan perbedaan tingkat metilasi (Hypo-DMG) dan gen dengan perbedaan tingkat transkrip (Up- dan Down-DEG). Hasilnya menunjukkan bahwa 372 gen mengalami penurunan tingkat metilasi dan peningkatan tingkat ekspresi; gen-gen ini ditetapkan sebagai gen hypo-up (Gambar 2g ). Anotasi Gene Ontology (GO) menunjukkan bahwa gen hypo-up terlibat dalam fungsi molekuler, seperti aktivitas regulasi transkripsi, aktivitas transpor, dan aktivitas antioksidan, dan dalam proses biologis, seperti proses reproduksi, proses ritmik, dan proses metabolisme (Gambar 2h ). Lebih jauh lagi, sebagai tambahan terhadap perbedaan signifikan dalam waktu pembungaan, galur WT dan epilate juga menunjukkan perbedaan signifikan dalam biomassa selama pertumbuhan vegetatif (Gambar S1a,b ) serta dalam kandungan klorofil (Gambar S1c ) dan terpenoid volatil (Gambar S1d ) pada daun. Karena 5-AzaC memiliki efek acak dan meluas pada tingkat metilasi DNA global, berbagai variasi fenotipik berbeda diharapkan terjadi.
CmFDa menghambat pembungaan pada krisan
Untuk lebih jauh mengeksplorasi mekanisme pengaturan epigenetik yang beroperasi pada epimutan epilat , kami meneliti pembungaan galur WT dan epilat dalam kondisi cahaya yang berbeda (LD dan hari pendek [SD]) (Gambar 3a,b ). WT memulai pembungaan setelah 20 minggu perbanyakan vegetatif dalam kondisi LD dan SD, sedangkan tanaman epilat tidak. Selain itu, jumlah total daun pada tanaman epilat secara signifikan lebih banyak daripada pada tanaman WT dalam kondisi SD dan LD (Gambar 3c,d ), yang mencerminkan fenotipe pembungaan tertunda dari epilat . Dengan demikian, epimutan epilat menunjukkan fenotipe pembungaan tertunda dalam kondisi LD dan SD.
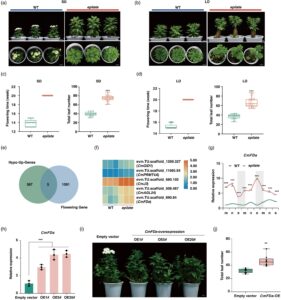
Untuk mengidentifikasi gen kunci yang bertanggung jawab atas fenotipe pembungaan tertunda pada epilat , kami mengidentifikasi 1096 gen homolog yang terkait dengan pembungaan dalam genom krisan (Bouché et al ., 2016 ; Liu et al ., 2024 ). Selanjutnya, kami memotong 1096 homolog gen terkait pembungaan ini dengan 372 CHH-Hypo-Up-Genes, yang menghasilkan lima Hypo-Up-Genes terkait pembungaan yang teregulasi naik (Gambar 3e ): evm.TU.scaffold_1269.327 ( CmGID1 ), evm.TU.scaffold_11665.94 ( CmPRMT4A ), evm.TU.scaffold_508.467 ( CmAGL24 ), evm.TU.scaffold_680.102 ( CmJ3 ) dan evm.TU.scaffold_690.84 ( CmFDa ) (Gambar 3f ). Semua gen ini berfungsi sebagai pengatur positif pembungaan (Ariizumi et al ., 2008 ; Niu et al ., 2008 ; Shen et al ., 2011 ); namun, beberapa laporan menunjukkan bahwa ekspresi berlebihan homolog FD dapat menghambat pembungaan pada spesies tertentu (Tylewicz et al ., 2015 ). Kami mengidentifikasi dua homolog FD dalam genom krisan: CmFDa ( evm.TU.scaffold_690.84 ), CmFDa-2 ( evm.TU.scaffold_11883.97 ) dan padanannya CmFDb ( evm.TU.scaffold_192.32 ) dan CmFDb-2 ( evm.TU.scaffold_61.93 ). Urutan asam amino FD dari spesies eukariotik yang berbeda diunduh dari basis data NCBI, dan pohon filogenetik dibangun menggunakan metode kemungkinan maksimum. Hasilnya menunjukkan bahwa protein FD dari Chrysanthemum seticuspe ( CsFD ) dan Artemisia annua ( AaFD ) paling dekat hubungannya dengan CmFDa (Gambar S2a ), dengan kesamaan urutan sekitar 97% (Gambar S2b ). Dibandingkan dengan urutan FD dari Arabidopsis, CmFDa tidak memiliki 100 asam amino N-terminal (Gambar S2b ). Nilai FPKM menunjukkan bahwa hanya ekspresi CmFDa yang berbeda secara signifikan antara garis WT dan epilat (Gambar S2c ). Hasil RT-qPCR mengungkapkan bahwa ekspresi CmFDa secara signifikan lebih tinggi pada garis epilat.daripada di WT (Gambar 3g ). Hal ini menunjukkan bahwa CmFDa dilestarikan dalam genus Chrysanthemum dan mungkin terlibat dalam mekanisme pengaturan pembungaan yang unik pada krisan.
Untuk memahami sepenuhnya peran CmFDa dalam pembungaan krisan, kami menghasilkan galur CmFDa overekspresi yang stabil ( CmFDa-OE ) pada latar belakang WT. Jumlah mRNA CmFDa meningkat secara signifikan pada tiga galur CmFDa overekspresi (Gambar 3h ). Galur CmFDa-OE berbunga lebih lambat daripada kelompok kontrol (EV, vektor Kosong) (Gambar 3i ). Kami menghitung jumlah daun pada keturunan yang diperbanyak secara aseksual dari galur CmFDa -OE dan EV. Hasilnya menunjukkan bahwa tanaman CmFDa overekspresi memiliki jumlah daun yang lebih banyak (Gambar 3j ). Selain itu, tanaman transgenik CmFDa-RNAi dengan latar belakang epilat memiliki jumlah daun yang lebih sedikit daripada tanaman epilat (Gambar S3a–c ), yang menunjukkan bahwa CmFDa memainkan peran penting dalam transisi bunga.
Ekspresi CmFDa yang lebih tinggi pada garis epilat dikaitkan dengan tingkat metilasi DNA yang lebih rendah pada promotornya
Untuk menyelidiki lebih lanjut kontribusi potensial dari epi-mutasi CmFDa terhadap ekspresi diferensialnya antara galur WT dan epilat , kami mengamplifikasi badan gen CmFDa dan daerah promoter (3000 bp hulu dari situs awal transkripsi [TSS]) dari galur WT dan epilat (Gambar 4a ). Kloning dan sekuensing fragmen yang diamplifikasi tidak mengungkapkan perbedaan dalam sekuens nukleotidanya antara kedua galur (Gambar S4 ). Namun, perbandingan dengan sekuens genom krisan mengungkapkan penyisipan sekitar 1000 bp di daerah promoter CmFDa (-1437 hingga -371 bp relatif terhadap TSS) (Gambar S4 ). Prediksi TE menggunakan perangkat lunak CENSOR ( http://www.girinst.org/censor/index.php ) mengungkapkan lima TE dalam promotor CmFDa , termasuk satu retrotransposon non-LTR, satu retrotransposon LTR dan masing-masing satu transposon DNA piggyBac, EnSpm dan MuDR (Tabel S2 ).
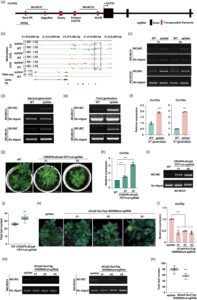
Bahasa Indonesia: Setelah koreksi, cuplikan layar IGV mengungkapkan bahwa tingkat metilasi DNA di wilayah promoter CmFDa yang membentang dari -3000 hingga -2000 bp secara komparatif lebih rendah pada epilat dibandingkan pada WT (Gambar 4b ). Mengingat latar belakang genetik yang identik dari kedua galur ini, kami selanjutnya menyelidiki tingkat metilasi DNA di dua wilayah spesifik promoter CmFDa (-2811 hingga -2013 bp dan -869 hingga -235 bp) pada WT dan epilat menggunakan McrBC-PCR (Gambar 4a ). Tingkat metilasi DNA di wilayah promoter CmFDa ini ditemukan lebih rendah pada epilat dibandingkan pada WT (Gambar 4c ), yang menunjukkan bahwa ekspresi CmFDa yang lebih tinggi pada epilat kemungkinan disebabkan oleh demetilasi promoternya. Selanjutnya, untuk mengeksplorasi apakah status metilasi promotor CmFDa dapat diwariskan secara mitosis, kami memeriksa tingkat ekspresi CmFDa dan status metilasi promotor CmFDa pada tanaman generasi kedua dan ketiga dari galur WT dan epilat (Gambar 4d–f ). Hasil penelitian menunjukkan bahwa perbedaan tingkat ekspresi gen dan tingkat metilasi promotor CmFDa diwariskan secara stabil oleh keturunan galur WT dan epilat .
Untuk lebih memvalidasi temuan tersebut di atas, kami menggunakan sistem demetilasi CRISPR-dCas9-TET1cd untuk menginduksi demetilasi DNA di daerah promoter CmFDa di galur WT. Enam single-guide RNA (sgRNA) dirancang untuk mengarahkan protein fusi dCas9-TET1cd ke promoter CmFDa , yang menghasilkan demetilasi signifikan promoter CmFDa di galur WT, yang mengarah pada peningkatan tingkat ekspresi CmFDa dan penundaan pembungaan (Gambar 4g–i ). Kami menghitung jumlah daun pada keturunan yang diperbanyak secara aseksual dari tanaman transgenik WT dan dCas9-TETcd-sgRNA . Hasilnya mengungkapkan bahwa tanaman transgenik dCas9-TETcd-sgRNA memiliki jumlah daun yang lebih tinggi (Gambar 4j ). Lebih jauh, kami menggunakan sistem metilasi dCas9-SunTag-NtDRM2cd untuk menginduksi metilasi DNA pada promotor CmFDa pada galur epilat . Tiga galur hasil suntingan genom diperoleh, yang semuanya menunjukkan peningkatan kadar metilasi DNA pada promotor CmFDa , penurunan ekspresi CmFDa , dan pembungaan awal dibandingkan dengan galur epilat yang tidak mengalami transformasi (Gambar 4k–m ). Perbandingan kuantitatif jumlah daun total antara tanaman generasi kedua yang dihasilkan oleh reproduksi aseksual galur transgenik WT dan dCas9-SunTag-NtDRM2cd menunjukkan bahwa tanaman transgenik dCas9-SunTag-NtDRM2cd memiliki jumlah daun yang lebih banyak (Gambar 4n ).
CmFDa bekerja sama dengan PRC2 untuk mengatur ekspresi gen promotor bunga
Untuk menyelidiki lebih lanjut mekanisme dasar yang bertanggung jawab atas keterlambatan pembungaan pada garis epilat , kami memilih daun apikal dari garis WT dan epilat untuk analisis transkriptom. Dengan memotong gen terkait pembungaan dengan set gen yang diatur turun (down-DEG), kami mengidentifikasi 28 gen pembungaan terkait down-DEG pada garis epilat (Gambar 5a ). Khususnya, ekspresi CmSOC1a dan CmAP1a secara signifikan diatur turun pada garis epilat (Gambar 5b ). Hasil RT-qPCR menunjukkan bahwa ekspresi CmSOC1a dan CmAP1a secara signifikan lebih rendah pada garis epilat dibandingkan pada garis WT (Gambar 5c ).
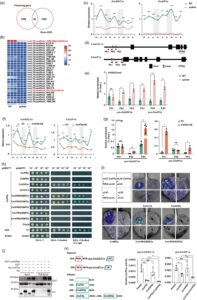
Untuk menyelidiki apakah ekspresi CmSOC1a dan CmAP1a berada di bawah regulasi epigenetik, kami mengukur kadar H3K27me3 dalam promotornya di garis WT dan epilate menggunakan ChIP-qPCR. Urutan promotor CmSOC1a (2136 bp hulu TSS) dan CmAP1a (675 bp hulu TSS) dikloning (Gambar 5d ). Dibandingkan dengan garis WT, peningkatan signifikan kadar H3K27me3 diamati di daerah promotor CmSOC1a dan CmAP1a di garis epilate (Gambar 5e ). Ini menunjukkan bahwa modifikasi histon H3K27me3 di daerah promotor CmSOC1a dan CmAP1a memainkan peran penting dalam menekan pembungaan.
Hasil RT-qPCR menunjukkan bahwa ekspresi CmSOC1a dan CmAP1a lebih rendah pada galur CmFDa-OE dibandingkan pada galur WT (Gambar 5f ). Untuk memverifikasi lebih lanjut bahwa CmFDa secara langsung mengatur ekspresi CmSOC1a dan CmAP1a , kami melakukan eksperimen ChIP menggunakan galur transgenik CmFDa-OE dan antibodi anti-FLAG. Hasil penelitian menunjukkan bahwa CmFDa dapat secara langsung mengikat situs PS2 dan PS3 pada promotor CmSOC1a dan situs PA1 dan PA3 pada promotor CmAP1a (Gambar 5g ).
Kami berhipotesis bahwa variasi kadar H3K27me3 dalam daerah promoter CmSOC1a dan CmAP1a antara garis WT dan epilate akan disebabkan oleh efek sinergis CmFDa dan PRC2. Untuk memvalidasi hipotesis ini, pertama-tama kami mengidentifikasi gen krisan yang mengkode komponen inti PRC2 (Tabel S3 , Gambar S5 , S6 ), dan kemudian memeriksa interaksi antara komponen CmFDa dan PRC2 menggunakan uji pencitraan komplementasi (LCI) ragi dua-hibrida (Y2H) dan luciferase (LUC). Dalam uji Y2H, CmFDa menunjukkan interaksi kuat dengan CmSWNa, CmVRN2/EMF2a dan CmVRN2/EMF2b, dan interaksi lemah dengan CmFIEa dan CmMSIa (Gambar 5h ). Interaksi CmFDa dengan PRC2 divalidasi melalui uji LCI pada daun tembakau ( Nicotiana benthamiana ) (Gambar 5i ). Untuk memeriksa lebih lanjut interaksi langsung antara CmFDa dan CmSWNa, kami memurnikan protein rekombinan, termasuk GST-CmSWNa, His-CmFDa dan His, serta GST. Percobaan pull-down lebih lanjut mengungkapkan bahwa CmFDa secara langsung mengikat CmSWNa secara in vitro (Gambar 5j ).
Berikutnya, kami melakukan uji reporter dual-LUC untuk menyelidiki efek penghambatan kompleks CmFDa–CmSWNa pada aktivitas promotor gen CmSOC1a dan CmAP1a (Gambar 5k ). Hasilnya menunjukkan bahwa CmFDa secara signifikan menekan aktivitas aktivasi transkripsi promotor CmSOC1a dan CmAP1a , sedangkan CmSWNa tidak berdampak. Efek penghambatan CmFDa pada promotor CmSOC1a dan CmAP1a selanjutnya ditingkatkan oleh fusinya dengan CmSWNa. Bersama-sama, hasil ini menunjukkan bahwa CmFDa berinteraksi dengan PRC2 untuk menekan ekspresi CmSOC1a dan CmAP1a , sehingga menghambat pembungaan.
Diskusi
Studi kami menyarankan model mekanistik untuk regulasi epigenetik pembungaan yang dimediasi CmFDa pada krisan. Demetilasi promotor gen CmFDa (dicapai menggunakan 5-AzaC dalam studi ini) menghasilkan peningkatan regulasi ekspresi CmFDa . CmFDa selanjutnya merekrut PRC2 ke promotor gen CmSOC1a dan CmAP1a , yang menyimpan modifikasi H3K27me3. Hal ini pada akhirnya mengarah pada penekanan ekspresi CmSOC1a dan CmAP1a dan akibatnya penundaan pembungaan (Gambar 6 ). Spesies tanaman yang berbeda dapat menggunakan mekanisme pensinyalan yang berbeda untuk merespons faktor endogen dan lingkungan yang sama yang mendorong pembungaan, yang menunjukkan keragaman fungsional dalam jalur pembungaan angiospermae. Misalnya, gandum, barley dan Arabidopsis memerlukan vernalisasi (perlakuan dingin yang berkepanjangan) untuk berbunga, sedangkan krisan tidak. Krisan merespons perubahan fotoperiode untuk berbunga. Di antara semuanya, semua jalur pensinyalan bunga diintegrasikan oleh gen FT dan SOC1 , yang pada akhirnya mengatur ekspresi gen identitas meristem bunga AP1 untuk mendorong pembungaan
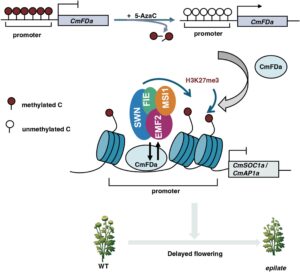
Bukti yang semakin banyak menunjukkan bahwa modifikasi histon memainkan peran penting dalam proses pembungaan. Meskipun banyak penelitian menunjukkan bahwa modifikasi epigenetik terlibat dalam pengaturan pembungaan pada tanaman, gen yang bertanggung jawab untuk menerjemahkan tanda epigenetik menjadi perilaku pembungaan masih harus dikarakterisasi sepenuhnya. H3K27me3 adalah tanda histon penghambat yang mengatur banyak proses perkembangan, termasuk yang menentukan waktu pembungaan. Penelitian sebelumnya telah difokuskan pada peran gen FLC dan FT dalam pembungaan (He et al ., 2020 ). Di sini, kami menunjukkan bahwa modifikasi H3K27me3 memengaruhi ekspresi gen pembungaan CmSOC1a dan CmAP1 a, yang konsisten dengan analisis genomik H3K27me3 di Arabidopsis (Del Olmo et al ., 2016 ). Namun, mekanisme yang mendasarinya belum dijelaskan secara rinci. Dilaporkan bahwa kompleks NF-Y mengikat promotor SOC1a melalui subunit NF-YA2 dan mengatur pengendapan H3K27me3, sebagian melalui demetilase H3K27 REF6, untuk mempengaruhi pembungaan (Cao et al ., 2014 ); namun, mekanisme yang bertanggung jawab untuk peningkatan level H3K27me3 masih belum diketahui. Kami menunjukkan bahwa penghambatan ekspresi CmSOC1a dan CmAP1a mungkin terkait dengan modifikasi histon H3K27me3, karena peningkatan level H3K27me3 di daerah promotor CmSOC1a dan CmAP1a , yang dicapai melalui perekrutan PRC2 oleh CmFDa, menghambat pembungaan pada garis krisan epilat . Ini menunjukkan bahwa FD sangat penting untuk pembungaan.
Dalam studi ini, ekspresi berlebihan CmFDa mengakibatkan penundaan yang signifikan dalam pembungaan, memberikan wawasan baru ke dalam regulasi epigenetik waktu pembungaan oleh CmFDa pada krisan. Protein FD dan protein mirip FD memainkan peran penting dalam regulasi transisi bunga pada angiospermae. Protein FD mengandung domain bZIP dengan afinitas pengikatan DNA yang tinggi. Kompleks TFL1–FD dilaporkan menghambat ratusan gen, termasuk yang terlibat dalam berbagai jalur pensinyalan endogen (termasuk sinyal gula dan hormon) (Zhu et al ., 2020 ). Dalam studi ini, kami menemukan bahwa selain perbedaan fenotipik dalam waktu pembungaan, galur WT dan epilat juga menunjukkan perbedaan yang signifikan dalam biomassa, klorofil daun, dan kandungan zat volatil. Kami mengidentifikasi DEG antara galur WT dan epilat melalui analisis transkriptom. Analisis pengayaan GO menunjukkan bahwa DEG ini berpartisipasi dalam berbagai proses biologis, seperti perkembangan, pertumbuhan, proses metabolisme, reproduksi, respons terhadap rangsangan eksternal, ritme dan pensinyalan, dan menjalankan berbagai fungsi molekuler seperti pengikatan dan regulasi transkripsi. Hal ini menunjukkan bahwa CmFDa tidak hanya mengatur pembungaan tetapi juga terlibat dalam proses lain yang terkait dengan pertumbuhan, perkembangan, dan regulasi metabolisme. Kompleks FD-PRC2 dapat menargetkan gen tambahan yang terlibat dalam regulasi negatif perkembangan tanaman dan metabolisme sekunder.
Dengan perkembangan teknologi penyuntingan genom tanaman, mengubah status metilasi DNA di situs genom tertentu untuk meningkatkan sifat tanaman telah menjadi sangat layak. Namun, menentukan urutan target yang akurat untuk menerapkan alat penyuntingan epigenom masih menjadi tantangan. Krisan memiliki genom besar dengan lebih dari 78% TE, yang dapat menjelaskan tingkat metilasinya yang relatif tinggi (Song et al ., 2023 ). Oleh karena itu, krisan merupakan material yang sangat baik untuk mempelajari regulasi metilasi DNA. Kami menganalisis karakteristik metilasi genom utuh dari krisan yang dibudidayakan secara poliploid, dan juga mempelajari fluktuasi tingkat metilasi DNA pada garis epilat , epimutan yang dihasilkan menggunakan 5-AzaC dan diidentifikasi berdasarkan fenotipe pembungaannya yang terlambat. Kami menemukan bahwa penghilangan tanda metilasi dari promotor CmFDa menyebabkan pembungaan tertunda pada krisan, dan perbedaan metilasi DNA pada promotor CmFDa dapat diwariskan pada tanaman WT dan epilate selama tiga generasi. Epilate secara konsisten menunjukkan pembungaan tertunda secara signifikan dibandingkan dengan WT. Kemudian, kami berhasil menggunakan sistem modifikasi epigenetik CRISPR-dCas9-TET1cd atau dCas9-SunTag-NtDRM2cd untuk mendemetilasi atau meningkatkan promotor CmFDa , yang mengakibatkan pembungaan tertunda atau lebih awal. Pertama kali ditunjukkan bahwa CmFDa adalah epialel baru yang mengatur pembungaan dan berfungsi sebagai contoh keadaan epigenetik yang dapat diwariskan. Ia dapat dipilih sebagai fenotipe klasik dalam program pemuliaan untuk mengendalikan waktu pembungaan krisan dengan mengubah ekspresi gennya.
Singkatnya, kami telah menyelidiki dasar epigenetik dari pengaturan waktu pembungaan pada krisan. Kami mengidentifikasi peran baru FDa dalam memediasi pengaturan gen hilir serta mekanisme aksi baru untuk PRC2 dalam pengaturan waktu pembungaan. Penggunaan 5-AzaC memungkinkan pembuatan epimutan non-transgenik, yang dapat membantu dalam penjelasan mekanisme dan meletakkan dasar untuk meningkatkan kinerja tanaman di lingkungan tertentu. Selain itu, ketika digunakan dalam kombinasi dengan sistem epimerase CRISPR-dCas9, epimutan ini dapat digunakan untuk validasi gen fungsional dan pemuliaan yang ditargetkan. Strategi ini memberikan pendekatan baru dan efektif untuk menciptakan keragaman epigenetik baru pada spesies tanaman.
Metode
Bahan tanaman
Aksesi krisan yang dibudidayakan ( Chrysanthemum morifolium , kultivar krisan penutup tanah) digunakan dalam penelitian ini. Bibit ditanam di tanah pada suhu 22 °C dalam kondisi LD (16 jam terang/8 jam gelap) atau SD (8 jam terang/16 jam gelap) di Institut Penelitian Bunga, Fakultas Ilmu Hayati, Universitas Kehutanan Timur Laut, Tiongkok.
Pengobatan 5-Azacytidine (5-AzaC)
Zat kimia 5-AzaC dilarutkan dalam dimetil sulfoksida (DMSO) dan kemudian ditambahkan ke 1× Fosfat Buffer Saline (PBS) pada konsentrasi akhir 50 μM. Untuk meningkatkan efek penetrasi zat terlarut, 0,05% (v/v) surfaktan L-77 ditambahkan ke larutan yang disiapkan. Kuncup kaki krisan dikumpulkan dari tanaman, dibersihkan di bawah air mengalir selama 1 jam dan ditempatkan dalam larutan yang disiapkan. Infiltrasi vakum dilakukan seperti yang dijelaskan sebelumnya. Secara singkat, pompa vakum digunakan untuk menerapkan vakum selama 15 menit, setelah itu udara dilepaskan dengan cepat. Proses ini diulang tiga kali untuk memungkinkan penghambat epigenetik sepenuhnya menembus jaringan kuncup. Setelah perawatan, kuncup kaki ditempatkan dalam keranjang pasir dan diinkubasi dalam gelap selama 24 jam. Selanjutnya, kuncup kaki dipindahkan ke kondisi terang dan dirawat lagi setelah 7 hari. Setelah kuncup menumbuhkan akar, mereka ditransplantasikan ke keranjang tanah.
Konstruksi plasmid dan transformasi tanaman
Untuk membuat vektor pCAMBIA1305.1-35S::CmFDa-3 × FLAG , urutan pengkodean CmFDa (CDS) diperkuat dari cDNA WT menggunakan 2 × Phanta Max Master Mix (Vazyme, Nanjing, Tiongkok) dan pasangan primer FLAG-CmFDa-F-( Pst I)/FLAG-CmFDa-R-( Hind III) (Tabel S4 ). Vektor pCAMBIA1305.1-35S-3 × FLAG dicerna dengan Pst I dan Hind III, dan CDS CmFDa dikloning ke dalam vektor pCAMBIA1305.1-35S-3 × FLAG yang dilinearisasi menggunakan ClonExpress II One Step Cloning Kit (Vazyme).
Untuk membangun knock down CmFDa dengan vektor RNAi, produk pemurnian fragmen maju dari CmFDa disisipkan berdekatan dengan situs pengenalan Nco I dan Asc I dari plasmid pFGC5941 dan selanjutnya, produk pemurnian fragmen mundur dari CmFDa disisipkan berdekatan dengan situs pengenalan Xba I dan BamH I dari plasmid pFGC5941.
Untuk membangun vektor biner CRISPR–dCas9–TET1cd untuk demetilasi DNA yang ditargetkan, enam sgRNA dirancang untuk setiap fragmen promotor dan dikloning ke dalam vektor tulang punggung AtU6-18T dan AtU3b-18T. AtU6-sgRNA1, AtU3b-sgRNA2, AtU6-sgRNA3, AtU3b-sgRNA4, AtU6-sgRNA5, dan AtU3b-sgRNA6 yang dihasilkan diperkuat menggunakan primer spesifik sekuens (Tabel S4 ). Vektor 1300-CRISPR–dCas9-TET1cd (Tang et al ., 2022 ) dicerna dengan Xma I, dan enam fragmen sgRNA yang dimurnikan dikloning secara terpisah ke dalam vektor biner yang dilinearisasi untuk menghasilkan enam konstruksi CRISPR–dCas9-TET1cd-sgRNA .
Untuk membangun vektor biner dCas9-SunTag-NtDRM2cd-sgRNAs untuk metilasi DNA yang ditargetkan. AtU6-sgRNA1, AtU3b-sgRNA2, AtU6-sgRNA3, AtU3b-sgRNA4, AtU6-sgRNA5 dan AtU3b-sgRNA6 dikloning secara terpisah ke dalam vektor linierisasi dCas9-SunTag-NtDRM2cd yang telah dicerna terlebih dahulu dengan KpnI untuk menghasilkan enam konstruksi dCas9-SunTag-NtDRM2cd-sgRNAs , secara terpisah. Vektor dCas9-SunTag-NtDRM2cd adalah hadiah dari Steven Jacobsen (Addgene plasmid #119 554; http://n2t.net/addgene:119554 ; RRID:Addgene_119 554 (Papikian et al ., 2019 )).
Konstruksi akhir ditransformasikan ke dalam sel Agrobacterium tumefaciens GV3101, yang kemudian digunakan untuk mentransformasi tanaman WT dan mencabutnya menggunakan metode infiltrasi vakum. Infiltrasi vakum pada kuncup bunga krisan telah dilakukan seperti yang dijelaskan sebelumnya (Li et al ., 2025 ; Tang et al ., 2022 ).
Ekstraksi DNA dan sekuensing bisulfit seluruh genom
DNA genomik diekstraksi dari daun tanaman WT dan epilate berusia 2 bulan menurut metode CTAB (Aidlab, Beijing, Cina), mengikuti petunjuk pabrik. Jaringan daun tanaman WT dan epilate , digiling dalam nitrogen cair sebagai bagian dari prosedur ekstraksi DNA genomik, dikirim ke Shanghai Biozeron Biotechnology Co., Ltd (Shanghai, Cina) untuk perawatan bisulfit, konstruksi pustaka, dan pengurutan Illumina.
RAD-seq, dan pemanggilan SNP
Sampel DNA diekstraksi dari daun tanaman berumur 2 bulan dari delapan galur krisan (‘BY’, ‘DXYP’, ‘FD’, ‘FSQ’, ‘YX’, ‘ZT’, WT, epilate ) dikirim ke Shanghai Biozeron Biotechnology Co., Ltd (Shanghai, Tiongkok) untuk pengurutan Illumina. Pustaka DNA diurutkan pada platform Illumina HiSeq PE150, dan data yang valid dianalisis untuk mengidentifikasi penanda SNP. Kemudian, tag RAD dalam setiap sampel dibandingkan dan diklasifikasikan, dan tabel frekuensi tag RAD setiap individu diperoleh menurut kedalaman pengurutan setiap jenis tag (dari besar ke kecil). Selanjutnya, tag RAD setiap sampel dibandingkan untuk mengidentifikasi situs heterozigot dalam sampel. Kemudian, tag RAD dari sampel yang berbeda dibandingkan satu sama lain untuk mengidentifikasi SNP antar individu. Akhirnya, berdasarkan informasi tabel frekuensi tag RAD dan informasi perbandingan setiap tag RAD, hasil yang mungkin berasal dari daerah duplikat disaring, dan hasil genotipe penanda SNP populasi berkeyakinan tinggi diperoleh. Untuk mengeksplorasi latar belakang genetik sampel, analisis komponen utama (PCA) dilakukan menggunakan perangkat lunak GCTA 1.93.2.
Uji urutan polimorfik amplifikasi terbelah (CAPS)
Berdasarkan analisis data dan verifikasi eksperimental, SNP ditemukan pada situs restriksi Sfc I dan Hpa I, dan primer PCR dirancang (Tabel S4 ). Produk PCR dimuat pada gel agarosa, dan ukuran fragmen yang diamplifikasi ditentukan dengan elektroforesis. Sampel dengan lokus SNP menunjukkan pita dengan panjang yang berbeda, yang digunakan untuk menentukan apakah sampel krisan WT, epilat , dan lainnya memiliki latar belakang genetik yang sama atau berbeda.
Ekstraksi RNA, pengurutan transkriptom dan analisis ekspresi gen
Total RNA diekstraksi dari daun tanaman krisan menggunakan Reagen TRIzol (Cwbio, Taizhou, Tiongkok), dan 1 μg total RNA yang diisolasi ditranskripsi balik menggunakan PrimeScript RT Reagent Kit (Perfect Real Time) (TaKaRa, Kyoto, Jepang). Selanjutnya, RT-qPCR dilakukan pada LightCycler480 II (Roche, Penzberg, Jerman) dalam reaksi 15 μL yang mengandung 7,5 μL FS Universal SYBR Green Master (Roche), 6,3 μL cDNA yang diencerkan 10 kali lipat, dan 0,6 μL masing-masing primer maju dan mundur. Gen EF digunakan sebagai kontrol internal, dan tiga replikasi biologis dilakukan untuk setiap sampel.
Untuk menganalisis transkriptom krisan, pustaka cDNA diurutkan menggunakan Sistem Illumina HiSeq X Ten (Illumina, San Diego, CA) oleh Shanghai Biozeron Biotechnology Co., Ltd (Shanghai, Cina).
Pengujian McrBC-PCR
Analisis McrBC-PCR dilakukan seperti yang dijelaskan sebelumnya (Tang et al ., 2022 ). Secara singkat, DNA genom diekstraksi dari daun tanaman krisan generasi pertama, kedua, dan ketiga. Kemudian, 100 ng DNA genom setiap sampel diperlakukan dengan 20 unit endonuklease McrBC pada suhu 37 °C selama 1–2 jam. Primer dirancang untuk menganalisis status metilasi transposon melalui elektroforesis PCR menggunakan produk yang dicerna sebagai templat (Tabel S4 ).
ChIP-qPCR
Uji ChIP dilakukan seperti yang dijelaskan sebelumnya dengan sedikit modifikasi (Tang et al ., 2022 ). Kromatin diekstraksi dari daun tanaman. Uji ChIP dilakukan dengan antibodi anti-Flag (F1804; Sigma) dan anti-trimetil H3K27 (Ab6002; Abcam). Kemudian, analisis qRT-PCR dilakukan dengan menggunakan primer spesifik (Tabel S4 ), dengan CmUBQ sebagai gen kontrol. Setelah normalisasi ke Input, tingkat pengayaan dihitung. Analisis imunopresipitasi dan PCR waktu nyata dari berbagai kelompok tanaman diulang setidaknya tiga kali untuk memastikan keandalan hasil eksperimen.
Pengujian ragi hibrida dua (Y2H)
CmFDa dikloning dalam vektor pGBKT7 (Takara) melalui rekombinasi homolog menggunakan situs Eco RI dan Bam HI. Demikian pula, CmCLFa , CmFIEa , CmMSIa , CmSWNa , CmVRN2/EMF2a , CmVRN2/EMF2b , CmVRN2/EMF2c , CmVRN2/EMF2d , dan CmVRN2/EMF2i secara individual dikloning ke dalam vektor pGADT7 (Takara) menggunakan situs Pst I dan Eco RI. Vektor pGADT7 dan pGBKT7 yang kosong digunakan sebagai kontrol negatif. Kloning DNA rekombinan dikonfirmasi melalui sekuensing Sanger, dan vektor umpan dan mangsa yang dikonfirmasi ditransformasikan bersama ke dalam sel Y2HGold ragi ( Saccharomyces cerevisiae ) (Takara), sesuai dengan petunjuk pabrik.
Pengujian pencitraan komplementasi luciferase (LCI)
Pengujian LCI dilakukan seperti yang dijelaskan sebelumnya, dengan sedikit modifikasi (Chen et al ., 2008 ). CmCLFa , CmSWNa , CmMSIa , CmVRN2/EMF2a, dan CmVRN2/EMF2b masing-masing dikloning ke dalam vektor pCAMBIA-nLUC, dan CmFDa dikloning ke dalam vektor pCAMBIA-cLUC. Konstruksi dan EV ini (pCAMBIA-nLUC dan pCAMBIA-cLUC) ditransformasikan ke dalam strain A. tumefaciens GV3101 menggunakan metode beku-cair. Berbagai kombinasi dirancang (cLUC- CmFDa + PRC2-nLUC), dan bakteri yang membawa pCAMBIA-nLUC dan pCAMBIA-cLUC (atau turunannya) dicampur bersama dalam proporsi yang sama dan diinfiltrasikan ke dalam daun Nicotiana benthamiana . Setelah 24 jam inkubasi gelap, substrat fluoresensi (E1602; Promega) diaplikasikan pada daun, dan luminesensi LUC difoto menggunakan Sistem Pencitraan Chemiluminescence Tanon 5200 (Tanon, Shanghai, Cina).
Uji tarik-turun
Protein fusi berlabel GST dan berlabel His dimurnikan sebagaimana dijelaskan (Wang et al ., 2025 ). Sekitar 100 μg GST-Vector atau GST-CmSWNa diinkubasi dengan kontrol His, atau GST-Vector atau GST-CmSWNa diinkubasi dengan His-CmFDa dalam sistem buffer pengikat 1 mL (50 mM Tris pH 7,5, 100 mM NaCl, 0,25% Triton X-100, 35 mM β-ME) selama 4 jam pada suhu 4 °C. Selanjutnya, 40 μL glutathione agarose atau Ni agarose yang telah diseimbangkan sebelumnya ditambahkan, dan sampel diinkubasi bersama pada suhu 4 °C selama 2–4 jam dengan pengadukan lembut. Sampel kemudian disentrifugasi pada 200 g selama 5 menit, dicuci 5 kali, lalu dianalisis dengan western blot.
Uji reporter dual-luciferase sementara
Uji reporter LUC dilakukan seperti yang dijelaskan sebelumnya, dengan sedikit modifikasi (Chen et al ., 2022 ). CmSWNa dan CmFDa masing-masing dikloning ke dalam vektor pGreenII 62-SK untuk menghasilkan konstruksi efektor, sedangkan CmSOC1a dan CmAP1a dikloning ke dalam vektor pGreenII 0800 untuk menghasilkan konstruksi reporter. Konstruksi efektor dan reporter, bersama dengan plasmid kosong pGreenII 62-SK dan pGreenII 0800, ditransformasikan ke dalam strain A. tumefaciens GV3101 menggunakan metode beku-cair. Serangkaian kombinasi unik dirancang, dan bakteri yang mengandung pGreenII 62-SK atau pGreenII 0800 (atau turunannya masing-masing) digabungkan dalam jumlah yang sama dan diinfiltrasikan ke dalam daun tanaman N. benthamiana berusia 4–5 minggu . Wilayah promotor (−2128 hingga −756 bp) dari gen CmSOC1a sepanjang 1373 bp dan (−675 hingga −1 bp) dari gen CmAP1a sepanjang 675 bp digunakan untuk menggerakkan gen LUC . Efek penghambatan CmFDa , CmSWNa, dan CmFDa-CmSWNa pada aktivitas promotor CmSOC1a dan CmAP1a dinilai secara kuantitatif dengan mengukur rasio aktivitas LUC terhadap aktivitas Renilla luciferase (REN), yang diukur menggunakan Dual-Luciferase Reporter Assay System (E1910; Promega).



Leave a Reply