Abstrak
Acibenzolar-S-methyl (ASM) adalah senyawa perlindungan tanaman yang dikenal untuk pengendalian bakteri, aktivasi tanaman, dan untuk meningkatkan toleransi stres panas dan kekeringan pada rumput turfgrass. Namun, respons transkripsi komprehensif terhadap perlakuan ASM pada creeping bentgrass (CB) belum dijelaskan. Untuk mengatasi hal ini, kami mengumpulkan pembacaan RNA-sequencing (RNA-seq) dari bentgrass untuk membuat set referensi gen yang diekspresikan. Dengan melakukan analisis RNA-seq pada sampel RNA dari daun yang diobati dengan ASM dan kontrol pada CB, kami mengidentifikasi secara komprehensif gen yang merespons perlakuan ASM. Untuk mengkarakterisasi sifat fungsional gen responsif ASM, kami menganalisis gen yang diduga mengkode faktor transkripsi (TF), termasuk WRKY TF. Kami mengidentifikasi gen yang diduga mengkode WRKY TF pada CB dengan respons yang dilestarikan terhadap ASM, analog dengan homolognya pada Brachypodium, yang diketahui memediasi pertahanan terhadap hawar seludang. Set gen responsif ASM yang teridentifikasi ini akan menyediakan sumber daya genomik yang berharga untuk analisis lebih rinci tentang ekspresi gen dan regulasi transkripsi sebagai respons terhadap pengobatan ASM di CB.
Singkatan
ASM
asam benzoat-S-metil
BTH
benzotiadiazol
Bahasa Inggris CB
rumput bengkok merambat
DEG
gen yang diekspresikan secara berbeda
PERGI
ontologi gen
Bahasa Indonesia: ORF
bingkai baca terbuka
PCA
analisis komponen utama
SA
asam salisilat
TF
faktor transkripsi
1. PENDAHULUAN
Creeping bentgrass (CB), Agrostis stolonifera L. (kultivar Penncross), dihargai di lapangan golf dan lapangan olahraga karena teksturnya yang halus, kepadatannya, dan toleransinya terhadap pemotongan yang rapat. Namun, sebagai rumput musim dingin, CB sangat rentan terhadap tekanan panas di luar kisaran optimalnya karena meningkatnya suhu permukaan (Morice et al., 2021 ), yang menyebabkan penurunan kualitas dan peningkatan kerentanan penyakit (Zhou et al., 2022 ). Khususnya, antraknosa (Young et al., 2008 ), pythium (Kerns & Tredway, 2007 ), bercak coklat (Toda et al., 2005 ), dan bercak dolar (Espevig et al., 2015 ) adalah penyakit rumput yang signifikan secara ekonomi, dan kejadiannya sangat sensitif terhadap kondisi cuaca (Kerns & Tredway, 2008 ), yang dapat berubah seiring dengan perubahan pola iklim. Lebih jauh lagi, Microdochium nivale , yang lazim di daerah beriklim sedang, menunjukkan respons yang berubah terhadap aktivator resistensi dengan kadar CO 2 yang bervariasi (Stricker et al., 2017 ). Aplikasi eksogen acibenzolar-S-methyl (ASM) telah ditunjukkan untuk meningkatkan toleransi panas dan kekeringan pada CB dengan memacu pertahanan metabolik melalui peningkatan senyawa terkait antioksidan (misalnya, prolin, serin, dan asparagin), serta meningkatkan osmolit dan metabolit terkait energi (Jespersen et al., 2017 ). Memahami respons fisiologis CB dalam kondisi yang terus berkembang ini sangat penting untuk mengembangkan strategi pemeliharaan berkelanjutan dan mengurangi biaya dalam menghadapi perubahan iklim.
Meskipun CB penting secara ekonomi, sumber daya genomiknya terbatas. Meskipun telah ada analisis transkriptom pada CB sebagai respons terhadap infeksi bercak dolar (Orshinsky et al., 2012 ) dan beberapa cekaman abiotik (Xu & Huang, 2018 ), koleksi profil transkriptom yang luas masih belum dieksplorasi. Sebaliknya, famili Poaceae, yang mencakup padi sebagai model monokotil yang dipelajari dengan baik, juga mencakup Brachypodium distachyon (Brachypodium), spesies rumput musim dingin model dalam Pooideae (Brkljacic et al., 2011 ; Hasterok et al., 2022 ), subfamili yang sama dengan CB. Studi pada Brachypodium, termasuk respons terhadap asam salisilat (SA) dan analognya ASM (juga dikenal sebagai benzothiadiazole [BTH]) (Kouzai, Noutoshi, et al., 2018 ), memberikan wawasan komparatif yang berharga. Analisis perbandingan semacam itu bermanfaat untuk meningkatkan pemahaman kita tentang mekanisme pertahanan dan meningkatkan strategi pengelolaan stres pada CB.
Dalam studi ini, kami menyelidiki efek ASM pada CB, terutama dalam kondisi lapangan. Sampel daun yang diolah dikumpulkan untuk RNA-sequencing (RNA-seq) untuk menyelidiki faktor transkripsi (TF) spesifik dalam keluarga besar regulator transkripsi dalam tanaman—TF WRKY mewakili TF dalam domain WRKY yang mengandung tanda tangan asam amino WRKY dekat N-terminus dan struktur zinc-finger baru dari C-terminus (Rushton et al., 2010 ). RNA-seq menghasilkan daftar lengkap gen yang diekspresikan, termasuk yang mengkode keluarga TF seperti WRKY. Selain itu, kami menilai pola ekspresinya sebagai respons terhadap perlakuan ASM dan mengidentifikasi TF WRKY yang responsif terhadap ASM. Hasil ini memfasilitasi analisis genomik komparatif dengan rumput model seperti Brachypodium , yang memberikan wawasan tentang mekanisme molekuler yang mendasari respons pertahanan yang diinduksi ASM pada CB.
2 BAHAN DAN METODE
2.1 Lokasi percobaan, aplikasi, dan pengumpulan jaringan daun
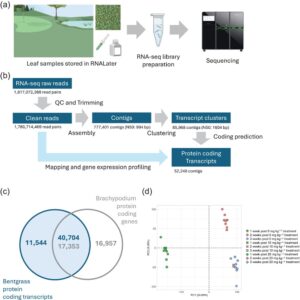
Uji coba lapangan dilakukan di Kawagoe Country Club (Saitama, Jepang, 36.089 LU, 139.383 BT) pada petak CB yang diberi ASM 10 dan 20 ppm pada tanggal 1 Juli 2021. Sampel daun dikumpulkan pada minggu ke-1, ke-2, dan ke-3 pasca-perlakuan (8 Juli–21 Juli 2021) pukul 13:00 dan langsung direndam dalam larutan RNAlater (Thermofisher Scientific) untuk mencegah degradasi RNA (Gambar 1a ). Tiga ulangan sampel disiapkan untuk setiap

2.2 Sampel RNA
Sampel RNA untuk perakitan transkriptom de novo diperoleh dari uji coba lapangan yang dijelaskan di atas, termasuk perlakuan ASM (10 mg kg −1 dan 20 mg kg −1 ) dan kontrol yang tidak diberi perlakuan. Untuk profil ekspresi berbasis 3′ RNA-seq, sampel daun disiapkan dari ruang pertumbuhan dengan ASM 20 mg kg −1 dan kontrol yang tidak diberi perlakuan. Sampel daun dikumpulkan pada 3 minggu pasca perlakuan dengan tiga kali ulangan sampel. Total RNA diekstraksi menggunakan RNeasy Plant Mini Kit (QIAGEN) sesuai dengan petunjuk pabrik. Kemudian, kualitas RNA dinilai menggunakan Agilent TapeStation (Agilent).
2.3 Pengurutan RNA
Dalam RNA-seq untuk perakitan transkriptom de novo, pustaka disiapkan menggunakan NextFlex Stranded RNA-seq Kit (Bioo Scientific) sesuai dengan petunjuk pabrik. Pengurutan dilakukan pada platform MGI DNBSEQ-T7 (MGI Tech Co., Ltd.) menggunakan metode paired-end (Gambar 1a ). Untuk pembuatan profil ekspresi sampel yang diberi dan tidak diberi ASM, pustaka pengurutan dibuat menggunakan QuantSeq 3′ mRNA-seq (Lexogen GmbH), dan pengurutan dilakukan pada sequencer Ion Proton (Thermofisher Scientific). Pembacaan urutan yang diperoleh dirangkum dalam Tabel S1 . Pembacaan mentah, setelah menghapus indeks dan adapter lainnya, dapat diakses melalui NCBI SRA dengan nomor akses berikut: PRJNA1254156.
2.4 Perakitan dan anotasi de novo
Pembacaan RNA-seq mentah diproses menggunakan Trimmomatic (v0.39) (Bolger et al., 2014 ) untuk memangkas basa berkualitas rendah dan membuang sekuens adaptor. Pembacaan berkualitas tinggi ini disusun menjadi kontig menggunakan Trinity (v2.8.5) (Grabherr et al., 2011 ) untuk perakitan transkriptom de novo. Kontig yang disusun kemudian dikelompokkan dengan CD-HIT-EST (v4.8.1) (Fu et al., 2012 ) untuk mengidentifikasi transkrip representatif, mengurangi redundansi, dan mengidentifikasi sekuens unik. Sekuens pengkodean dalam transkrip representatif diprediksi menggunakan TransDecoder (v5.5.0) ( https://github.com/TransDecoder ) (Gambar 1b ). Set transkrip pengkode protein yang diduga diberi anotasi fungsional menggunakan pencarian homologi berbasis BLAST dengan set data protein termasuk Swissport dan proteom yang disimpulkan dalam genom Arabidopsis , padi, dan Brachypodium (Tabel S2 ). Anotasi ontologi gen (GO) untuk analisis pengayaan GO dilakukan menggunakan metodologi anotasi fungsional Blast2GO di OmicsBox (Götz et al., 2008 ).
2.5 Profil ekspresi
Pembacaan yang dipangkas kemudian dipetakan ke set transkrip pengodean protein putatif menggunakan Hisat2 (Kim et al., 2019 ) dan tmap (v3.0.1, https://github.com/nh13/TMAP ) untuk pembacaan yang diperoleh dari MGIseq dan sequencer IonProton, masing-masing, dan featureCounts (v2.0.1) (Liao et al., 2014 ) digunakan untuk mendapatkan data jumlah pembacaan untuk setiap transkrip. Untuk membandingkan tingkat ekspresi transkrip yang relevan, pembacaan per juta (RPM) digunakan. Gen yang diekspresikan secara berbeda (DEG) dipilih menggunakan DESeq2 (Love et al., 2014 ). Uji pengayaan GO dilakukan dengan paket TopGO R (Alexa & Rahnenfuhrer, 2024 ), dan signifikansi statistik dinilai menggunakan uji pasti Fisher.
3 HASIL DAN PEMBAHASAN
Melalui RNA-seq, kami memperoleh total 1.817.072.388 bacaan mentah, yang dipangkas menjadi 1.780.714.469 bacaan berkualitas tinggi, dengan rata-rata 65 juta bacaan per sampel. Ini merupakan salah satu upaya pengurutan terdalam untuk RNA-seq CB hingga saat ini. Kami menghasilkan 777.401 kontig dengan N50 994 bp, dan selanjutnya menghasilkan 146.723 super-kontig dengan N50 1547 bp. Pengelompokan super-kontig menghasilkan 85.968 klaster dengan N50 1604 bp (Gambar 1b ). Dari jumlah ini, 52.248 kontig representatif diprediksi mengandung kerangka baca terbuka (ORF), yang membentuk set transkrip referensi kami (Gambar 1b ). Dari 52.248 ORF yang diprediksi dalam CB, 40.740 menunjukkan kesamaan yang signifikan dengan 17.353 gen dalam genom B. distachyon (Gambar 1c , Tabel S2 ). Ini termasuk 1782 transkrip yang diidentifikasi sebagai homolog dengan TF putatif dalam B. distachyon , yang sesuai dengan 958 gen TF nonredundan dalam B. distachyon (Tabel S2 ). Tingkat kesamaan yang tinggi ini menunjukkan tumpang tindih substansial dalam potensi pengkodean antara CB dan model rumput yang dipelajari dengan baik. Meskipun rakitan masih mengandung redundansi, itu mungkin disebabkan oleh poliploidi genom CB (MacBryde, 2006 ), yang kemungkinan berkontribusi pada duplikasi gen yang diamati dan variasi sekuens dari sekuens yang ditranskripsi di antara ortolog.
Pembacaan yang dipangkas dipetakan ke set transkrip pengode protein yang dibangun di atas untuk memperkirakan tingkat ekspresi gen untuk setiap sampel. Distribusi sampel berbasis jumlah pembacaan yang dinormalisasi direpresentasikan menggunakan analisis komponen utama (PCA). Hasil PCA menunjukkan variabilitas substansial di seluruh titik pengambilan sampel yang berbeda, dengan variabilitas ini lebih jelas daripada perbedaan yang disebabkan oleh ada atau tidak adanya perlakuan ASM (Gambar 1d ). Pengamatan ini menunjukkan bahwa profil ekspresi gen global CB yang tumbuh di lapangan sangat dipengaruhi oleh kondisi lingkungan dan keadaan fisiologis tanaman pada saat pengambilan sampel. Pengamatan penting adalah perbedaan signifikan dalam profil transkriptom antara minggu pertama dan minggu-minggu berikutnya (2 atau 3 minggu setelah perlakuan) bahkan dalam sampel kontrol. Ketika berfokus pada DEG yang berfluktuasi tergantung pada waktu pengambilan sampel, kami menemukan bahwa persentase gen yang cukup besar secara signifikan meningkat (5767 transkrip, 11,04%) atau menurun (5520 transkrip, 10,57%) pada minggu kedua atau ketiga dibandingkan dengan minggu pertama. Analisis pengayaan GO dari DEG ini mengungkap perubahan signifikan dalam proses biologis seperti pelipatan protein dan respons terhadap panas ( p < 1e-30, uji pasti Fisher) di antara transkrip yang diatur naik, sementara proses seperti fosforilasi ( p < 5.1e-25, uji pasti Fisher) dan fotosintesis, khususnya pemanenan cahaya dalam fotosistem I ( p < 8.3e-11, uji pasti Fisher), menonjol dalam transkrip yang diatur turun (Tabel S3 ). Ini menunjukkan bahwa gen ini menunjukkan fluktuasi yang cukup besar dalam CB yang tumbuh di lapangan.
Dengan menggunakan set transkrip pengkode protein, kami melakukan penelusuran homologi terhadap daftar TF yang diklasifikasikan berdasarkan domain pengikatan DNA pada Brachypodium dan padi. Dengan mengadopsi homolog terdekat, kami mengidentifikasi transkrip yang secara putatif mengkode TF melalui penelusuran homologi untuk daftar TF padi dan Brachypodium (Jin et al., 2017 ). Di antara ini, TF WRKY patut diperhatikan, dengan 89 transkrip yang diprediksi. Kami menyelidiki responsivitasnya terhadap perlakuan ASM. Kami menumpangkan tingkat ekspresi gen-gen ini sebagai respons terhadap SA dan BTH pada Brachypodium (Kouzai, Kimura et al., 2018 ; Kouzai, Noutoshi et al., 2018 ), dan perlakuan ASM pada CB (Tabel S4 ). Analisis ini memungkinkan kami untuk mengidentifikasi beberapa TF WRKY dengan respons yang dilestarikan terhadap BTH atau ASM dalam subfamili Pooideae. Dua gen responsif ASM yang diduga mengkode TF WRKY, Agrostis85463c000010 dan Agrostis52628c000010 , menunjukkan homologi tinggi dengan TF utama Bradi4g30360 dan Bradi2g22241 di Brachypodium , yang keduanya merupakan gen pengatur yang terkait dengan ketahanan terhadap penyakit hawar seludang (Kouzai et al., 2020 ). Temuan ini menunjukkan bahwa TF WRKY ini mungkin memainkan peran penting dalam respons pertahanan yang dimediasi ASM di CB. Gen responsif ASM ini, termasuk homolog TF WRKY, berpotensi sebagai penanda respons ASM, memberikan wawasan berharga ke dalam mekanisme molekuler yang mendasari respons pertahanan yang diinduksi ASM di CB.
Studi ini menyediakan kumpulan data transkriptom yang diurutkan paling dalam untuk daun CB hingga saat ini. Meskipun genom CB belum sepenuhnya diurutkan, set transkrip daun yang kami rakit berfungsi sebagai proksi genomik, yang menawarkan landasan yang berharga untuk menyelidiki ekspresi dan regulasi gen dalam spesies ini. Kumpulan data ini berkontribusi pada pemahaman yang lebih dalam tentang dinamika transkripsi dalam CB, khususnya dalam kondisi lapangan yang kompleks dan berfluktuasi. Temuan kami menunjukkan bahwa variasi transkripsi dalam CB yang tumbuh di lapangan sebagian besar dipengaruhi oleh faktor lingkungan seperti iklim dan manajemen. Selain analisis transkriptomik berbasis lapangan, kami mengidentifikasi TF WRKY yang mungkin terlibat dalam respons ASM. TF ini berpotensi sebagai penanda molekuler untuk melacak respons ASM di berbagai lingkungan serta target potensial untuk menyempurnakan pemahaman kita tentang tindakan ASM pada tingkat transkripsi. Dengan memanfaatkan kumpulan data ini, penelitian di masa mendatang dapat meningkatkan ketepatan analisis respons ASM dan berkontribusi untuk mengoptimalkan strategi pengelolaan rumput.



Leave a Reply