ABSTRAK
Cina membanggakan sumber daya genetik ayam lokal yang melimpah, di mana eksploitasi informasi polimorfisme nukleotida tunggal (SNP) genom utuh menawarkan potensi signifikan bagi pengembangannya. Namun, chip SNP ayam saat ini terutama dirancang untuk ayam komersial atau beberapa ras lokal. Untuk mengatasi kesenjangan ini, kami telah mengembangkan chip cair “Shennong 1 chicken 40K” yang memanfaatkan genotipe dengan pengurutan yang ditargetkan. Chip ini mengintegrasikan SNP dan varian yang ada/tidak ada dan secara khusus dibuat untuk ayam lokal Cina. Ini mencakup 44.849 situs target, yang dipilih melalui integrasi data pengurutan ulang genom utuh, data pan-genom, data studi asosiasi genom-lebar, dan data fungsional yang dilaporkan sebelumnya untuk sifat ekonomi. Dibandingkan dengan chip gen yang dipublikasikan, chip ini mengandung lebih banyak lokus polimorfik pada ayam lokal Cina, yang menunjukkan penerapan yang lebih baik. Validasi kami terhadap chip pada 204 individu dari tujuh ras berbeda menghasilkan rasio tangkapan rata-rata sebesar 99,474% untuk situs target, dengan frekuensi alel minor > 0,05 yang mencakup 98,557% dari total situs. Chip ini secara efektif mengklasifikasikan ras yang berbeda, menyelaraskan hasil pengelompokan dari analisis struktur populasi dengan pengelompokan ras yang sebenarnya, dengan demikian menunjukkan penerapan chip yang sangat baik. Selain itu, kami mengidentifikasi gen yang terkait dengan produksi dan adaptasi lingkungan pada ayam melalui analisis sinyal seleksi ( IGF1, SOX5, CACNA1G, dan CXCR4 ). Yang penting, situs fungsional chip memungkinkan evaluasi yang tepat, membantu dalam memahami sifat ekonomi ras tertentu untuk pengambilan keputusan yang tepat. Secara keseluruhan, chip memberikan dukungan teknis penting untuk konservasi, pembiakan, identifikasi, dan evaluasi sumber daya genetik ayam asli Tiongkok.
1 Pendahuluan
Bentang alam genetika Tiongkok yang luas, yang menjadi rumah bagi 115 ras ayam lokal, menghadapi tantangan mendesak dari serbuan ras komersial berproduksi tinggi, yang menyebabkan penurunan keragaman genetika dan keberadaan pasar ras lokal [ 1 , 2 ]. Tren ini tidak hanya menghambat produksi reguler tetapi juga mengancam pembiakan unggas yang berkelanjutan. Hambatan kritis adalah tidak adanya teknik konservasi tingkat genom dan sistem pembiakan dan evaluasi yang efisien dan hemat biaya. Oleh karena itu, pengembangan chip genom cair, yang disesuaikan dengan kebutuhan unik ayam lokal Tiongkok, sangat penting untuk memajukan penelitian genetika dan praktik pembiakan berkelanjutan. Chip polimorfisme nukleotida tunggal (SNP), yang terkenal karena throughput tinggi dan efektivitas biayanya, telah merevolusi teknologi genotipe dalam pembiakan ternak. Hal ini dibuktikan pada berbagai spesies, termasuk sapi [ 3 ], domba [ 4 ], dan babi [ 5 ], antara lain [ 6 , 7 ]. Dalam panel genotipe unggas, chip SNP 3K pertama pada tahun 2005 menandai tonggak penting [ 8 ], digantikan oleh susunan genotipe SNP Affymetrix 600K ayam [ 9 ]. Susunan ini telah menjadi sangat diperlukan dalam membedah hubungan genetik di antara ras hewan, memfasilitasi studi asosiasi genom-lebar (GWAS) [ 7 ], mendeteksi tanda-tanda seleksi dan pola evolusi, dan menilai keragaman genetik dan seleksi genom [ 10 ]. Khususnya, peluncuran chip SNP ayam pedaging 55K pada tahun 2017 [ 6 ] dan chip SNP ayam petelur 50K pada tahun 2018 [ 11 ] telah menjadi penting dalam mendorong pengembangan independen ayam komersial di Cina. Prinsip dasar yang mendasari chip ini melibatkan hibridisasi probe pada slide untuk membalikkan urutan komplementer dari situs target. Namun, biaya kustomisasi dan genotipe susunan ini tetap tinggi, menimbulkan tantangan dalam beradaptasi dengan berbagai skenario aplikasi. Mengingat ketergantungannya pada kaca slide sebagai pembawa, chip SNP ini disebut sebagai chip padat.
Kemajuan terkini telah menyaksikan munculnya teknik baru yang dikenal sebagai SNP-based genotyping by targeted sequencing (GBTS), sebuah lompatan signifikan dalam genomik [ 12 ]. Chip SNP yang dikembangkan berdasarkan teknologi GBTS umumnya dikenal sebagai “liquid chip” karena sistem reaksinya beroperasi dalam media cair. Chip cair menawarkan resolusi yang lebih tinggi, biaya yang lebih rendah, dan fleksibilitas yang lebih besar dibandingkan dengan chip padat. Awalnya diterapkan pada jagung, GBTS memfasilitasi pembuatan chip 20 dan 40K [ 12 ]. Susunan SNP berbasis GBTS telah dirancang untuk lebih dari 100 organisme, yang dicontohkan oleh chip cair kapas 40K “ZJU CottonSNP40K” [ 13 ]. Teknologi GBTS telah berperan penting dalam memperluas pemahaman kita tentang keragaman dan struktur genetik populasi [ 14 ], membantu dalam pemetaan lokus sifat kuantitatif (QTL), konstruksi peta genetik [ 13 ], GWAS, dan penambangan gen [ 15 ]. Keserbagunaannya dan cakupan aplikasinya yang luas dengan cepat menjadikan GBTS sebagai teknologi utama bagi berbagai bidang biologi. Ini termasuk evaluasi sumber daya genetik, lokalisasi dan kloning gen, konstruksi peta genetik, seleksi dengan bantuan penanda molekuler, konservasi hewan, pemantauan kualitas benih, evaluasi keamanan, pengujian biologis, dan banyak lagi [ 13 ].
Saat ini, hanya beberapa chip SNP 5K berdensitas rendah [ 16 ] atau desain spesifik breed regional yang tersedia untuk populasi ayam asli Tiongkok. Ada kebutuhan mendesak untuk chip tipe multivarian berdensitas sedang dengan cakupan genomik yang lebih luas dan representasi polimorfisme yang seimbang. Dengan menggabungkan situs pan-genom yang diidentifikasi sebelumnya [ 17 ], penampang yang lebih representatif dari seluruh sekuens spesies ayam dapat dicapai. Pendekatan ini meningkatkan kemampuan chip untuk menangkap variasi struktural, seperti varian yang ada/tidak ada (PAV) [ 18 ]. Hal ini mengarah pada cakupan informasi yang lebih komprehensif dan evaluasi yang lebih akurat. Oleh karena itu, pengembangan chip genom cair untuk ayam asli Tiongkok bukan hanya kemajuan dalam teknologi genotipe tetapi langkah maju yang penting dalam konservasi, pembiakan dan seleksi, identifikasi sumber daya, dan evaluasi sumber daya genetik ayam asli Tiongkok.
Dalam penelitian ini, kami mengembangkan chip genom cair, yang diberi nama “Shennong 1 chicken 40K chip,” dan menggunakannya untuk membuat genotipe 204 individu. Kemanjurannya dibuktikan melalui analisis genetik populasi yang komprehensif, yang mencakup evaluasi keragaman genetik, struktur populasi, riwayat populasi, dan analisis sinyal seleksi.
2 Bahan dan Metode
2.1 Pengumpulan Data Resequencing Genom Secara Keseluruhan
Untuk memilih lokasi target yang optimal untuk chip genom ayam cair, kami melengkapi set data ini dengan data genom utuh yang diunduh dari 226 individu dari 22 ras dari basis data Arsip Bacaan Urutan Pusat Informasi Bioteknologi Nasional (NCBI). Lebih jauh, kami mengumpulkan sampel DNA dari 418 ayam dari 36 ras asli menggunakan Kit DNA Genomik TIANamp (Tiangen Biotech Co. Ltd., Beijing, Tiongkok). Setelah melalui kontrol kualitas yang ketat, semua sampel menjalani pengurutan berpasangan (PE150) pada platform MGISEQ-500, yang bertujuan untuk mendapatkan kedalaman pengurutan rata-rata 10x per sampel. Dengan menggabungkan data ini, terdapat total 58 ras dan 647 individu. Rincian yang diberikan pada Gambar 1 dan Tabel S1 menjadi dasar analisis kami.
Pembacaan mentah disaring menggunakan fastp [ 19 ] dengan parameter default untuk menghilangkan urutan adaptor, basa berkualitas rendah, dan basa yang tidak ditentukan, sehingga menghasilkan pembacaan bersih berkualitas tinggi. Urutan 66,4 Mb baru yang tidak ada dalam genom referensi ayam GRCg6a ( https://www.ncbi.nlm.nih.gov/datasets/genome/GCF_000002315.6/ ) dari studi pan-genom sebelumnya [ 17 ] dimasukkan sebagai pseudokromosom ke dalam GRCg6a, sehingga menciptakan genom referensi augmented, disebut sebagai GRCg6a_pan_genome, untuk deteksi varian. Pemetaan pembacaan bersih ke GRCg6a_pan_genome dieksekusi menggunakan BWA-MEM [ 20 ], dan file penyelarasan diindeks, dan pembacaan duplikat dihilangkan melalui SAMtools v.1.40 [ 21 ]. Pemanggilan varian dilakukan dengan pemanggilan SNP yang dilakukan melalui jalur GATK. Terakhir, PLINK v1.9 [ 22 ] digunakan untuk menyaring situs dengan tingkat kehilangan yang tinggi dan frekuensi alel minor yang rendah (parameter: –geno 0.2, –maf 0.05), menghasilkan set SNP berkualitas tinggi sebanyak 12.381.606.
2.2 Pemilihan Lokasi Chip dari Calon Lokasi Target
Lokasi chip dibagi menjadi empat bagian, seperti yang diilustrasikan pada Gambar 2 :
- Situs dipilih dari 12.381.606 SNP menggunakan metode penilaian SNP. Untuk mencapai tujuan ini, SNP dengan skor tertinggi di setiap jendela (ukuran jendela 30 Kb dan langkah jendela 25 Kb) dipertahankan. Menurut rumus [ 23 ]: Skor = MAF × [(E − S) − |2a − (E + S)|], di mana MAF mewakili frekuensi alel minor, E dan S masing-masing mewakili koordinat akhir dan awal jendela, dan a menandakan koordinat SNP. Menurut rumus, skor SNP yang lebih tinggi berarti polimorfisme lokus yang lebih baik dan distribusi yang lebih merata.
- Penggabungan sekuens pan-genom memungkinkan chip untuk membuat genotipe PAV. Bersamaan dengan itu, perakitan sekuens pan-genom ini berasal dari data sekuensing ayam lokal dan komersial dari seluruh dunia. Oleh karena itu, informasi SNP pada sekuens ini, dibandingkan dengan genom referensi tunggal, mungkin memiliki potensi keragaman genetik. Oleh karena itu, kami memilih set kedua situs SNP kandidat dari sekuens pan-genom ini. Mengenai pemilihan kelompok ini, pertama-tama kami memilih dua situs per sekuens dalam pan-genom, baik dengan homologi kurang dari 5 atau dua situs dengan homologi terendah, ditambah dengan konten GC yang optimal. Jika semua situs yang dirancang dalam sekuens memiliki homologi lebih besar dari 5, kami akan menghentikan penggunaan sekuens itu dalam pan-genom.
- Dalam studi ini, pemilihan situs SNP dilakukan dengan menggunakan analisis GWAS dari 15 sifat, seperti yang digambarkan dalam Ref. [ 24 ]. Populasi ini berasal dari persilangan ayam Beijing You dengan ayam pedaging Cobb (Cobb-Vantress Inc.). Pengukuran fenotip dicatat untuk 367 individu dari generasi F2. Karena ketidakkonsistenan antara genom referensi yang digunakan dalam analisis GWAS ini dan referensi kami saat ini, kami kemudian mengumpulkan informasi lokus dan menggunakan alat liftOver [ 25 ] untuk memetakannya kembali ke genom referensi GRCg6a_pan_genome. Pemetaan ulang ini menghasilkan data lokus yang diperbarui. Untuk situs yang berhasil dipetakan, perangkat lunak perancang probe GenoBaits (Jianan Zhang, MolBreeding Biotech.) digunakan untuk evaluasi dan desain probe. Situs yang gagal dalam evaluasi ini dikeluarkan dari pertimbangan lebih lanjut.
- Untuk mencapai evaluasi yang tepat terhadap sifat-sifat khusus ras, kami secara ekstensif mengumpulkan situs mutasi kausal yang dilaporkan sebelumnya yang memengaruhi sifat-sifat produksi ekonomi utama pada ayam. Dengan memanfaatkan ketersediaan sekuens pan-genom, chip kami tidak hanya dapat mendeteksi SNP tetapi juga menangkap variasi struktural seperti PAV. Kumpulan situs ini mencakup lokus yang terkait dengan pertumbuhan, berat badan, warna bulu, warna kaki, warna daging, warna kulit, dan warna kulit telur (56 situs). Terakhir, kami memetakan semua situs ke genom referensi GRCg6a dan kemudian mengevaluasi kesesuaian sekuens flanking untuk desain probe menggunakan alat perancang probe GenoBaits.
-

Peta jalan dan karakterisasi lokasi target pada chip ayam Shennong 1 40K.
2.3 Evaluasi Probe dan Genotyping
Dalam metodologi kami untuk pemilihan lokasi SNP, desain probe mematuhi kriteria yang ketat: (1) panjang probe 110 bp, (2) kandungan GC berkisar antara 30% hingga 70%, dan (3) kurang dari lima daerah homolog. Alat perancang probe GenoBaits digunakan untuk menilai apakah lokasi SNP dalam tiga kelompok pertama memenuhi kriteria. Setelah sintesis dengan hasil tinggi, probe ini digunakan untuk GBTS.
DNA genomik diekstraksi dari darah utuh menggunakan TIANamp Genomic DNA Kit. Konstruksi perpustakaan dilakukan menggunakan GenoBaits DNA-seq Library Preparation Kit (MolBreeding Biotechnology Co., Shijiazhuang, Hebei, Tiongkok). Kemudian, probe dan buffer hibridisasi dicampur dan dihibridisasi pada 65°C selama 16 jam. Dynabeads MyOne Streptavidin C1 dan buffer pengikat ditambahkan ke volume untuk memperkaya fragmen DNA target dan membuang fragmen DNA nontarget. Fragmen target diperkuat menggunakan primer amplifikasi perpustakaan dan DNA polimerase, diikuti oleh dua putaran pemurnian dengan Beckman AMPure Beads. Akhirnya, konsentrasi perpustakaan diukur menggunakan Qubit 2.0 Fluorometer (Thermo Fisher Scientific, CA) dan qPCR. Pengurutan fragmen DNA target dilakukan pada platform MGISEQ-T7 (MGI, Shenzhen, Tiongkok), dengan pemanggilan SNP dilakukan melalui jalur GATK [ 26 ].
2.4 Perbandingan Shennong 1 Chicken 40K Chip dengan Chicken Arrays yang Ada
Untuk menilai secara komprehensif penerapan chip Shennong dan chip yang dipublikasikan untuk ras ayam asli Tiongkok, kami mengumpulkan lokus dari tiga chip gen utama yang saat ini tersedia di pasaran (Illumina 60K SNP BeadChip, susunan ayam Affymetrix 600K, dan chip Jingxin 1 55K v3_version) sebagai perbandingan. Perbandingan difokuskan pada tiga aspek utama: (1) menghitung proporsi situs polimorfik, yang mengukur penerapan situs chip pada ras; (2) mengevaluasi keseragaman distribusi lokus, yang menilai akurasi dan efektivitas analisis genomik; dan (3) melakukan analisis anotasi struktur genomik.
Analisis dilakukan sebagai berikut: menggunakan alat liftOver [ 27 ], kami mengonversi situs dari dua chip (Illumina 60K SNP BeadChip dan Affymetrix 600K chicken array) ke koordinat genom referensi GRCg6a. Berdasarkan situs dari setiap chip, kami memanggil kumpulan data variasi dari 58 ras ayam asli Tiongkok (total 644 individu) untuk memperoleh proporsi situs polimorfik dalam populasi ayam asli Tiongkok, yang memungkinkan perbandingan penerapan setiap chip. Evaluasi didasarkan pada situs polimorfik sebagai indikator, yang mendefinisikan situs nonpolimorfik sebagai situs di mana MAF populasi < 0,05 [ 6 ], karena pada situs tersebut, keadaan akan homozigot, tidak berdampak pada analisis. Semakin banyak situs nonpolimorfik, semakin tidak cocok chip untuk ras tersebut. Selain itu, untuk memvalidasi keandalan metode kami, kami menggunakan data genotipe susunan SNP ayam Affymetrix 600K untuk ayam lokal Tiongkok (8 ras, 157 individu) dari basis data publik [ 28 ], dengan melakukan perhitungan yang sama. Analisis anotasi struktur genom dari situs chip dilakukan dengan menggunakan perangkat lunak snpEff v5.1 [ 29 ].
2.5 Aplikasi Shennong 1 Chicken 40K Chip pada Lima Jenis Ayam
Kami menganalisis 204 individu dari tujuh ras ayam (enam ayam lokal Tiongkok dan satu ayam komersial) untuk memvalidasi efisiensi deteksi chip (Tabel 1 ). Ini termasuk ayam Lasa putih, ras yang dikembangkan dari persilangan ayam jantan White Leghorn dengan ayam betina Tibet, dan ayam Rhode Island Red, yang secara luas digunakan sebagai nenek moyang ayam petelur komersial bercangkang coklat. Ayam Lasa putih dan Rhode Island Red disertakan untuk mengeksplorasi efektivitas deteksi chip pada ras global lainnya, yang bertujuan untuk memperluas cakupan aplikasi chip ayam Shennong 1 40K.
| Ras | Label | Nomor | Jenis |
|---|---|---|---|
| Ayam Gushi | HGS | 33 | Jenis telur dan daging; dataran rendah |
| Ayam aduan Henan | HFC | 30 | Tipe suka bermain dan suka berfoya-foya; dataran rendah |
| Ayam tiga kuning Zhengyang | Bahasa Indonesia: ZYT | 30 | Jenis telur dan daging; dataran rendah |
| Ayam tulang hitam Xichuan | Sepeda XCB | 28 | Jenis obat dan makanan; dataran rendah |
| Ayam panggang | HLS | 30 | Jenis telur dan daging; dataran rendah |
| Lasa ayam putih | Akan segera diumumkan | 30 | Tipe telur; tipe adaptasi dataran tinggi |
| Ayam Rhode Island Merah | Bahasa Indonesia: RIR | 23 | Ayam komersial; dataran rendah |
Bahasa Indonesia: Setelah memperoleh data genotipe untuk semua individu, kami melakukan statistik tingkat penangkapan, MAF situs dan distribusi situs, dan analisis genetik populasi. Dalam analisis keragaman genetik sambil meminimalkan bias terkait kromosom seks, kami hanya berfokus pada SNP autosomal, mengecualikan 2540 SNP dari kromosom seks [ 30 ]. Lebih jauh, kami menggunakan PLINK v1.9 untuk menghitung heterozigositas yang diamati (Ho), heterozigositas yang diharapkan (He), MAF, dan koefisien perkawinan sedarah (F HOM ) dari populasi. Untuk analisis struktur populasi, kami melakukan analisis komponen utama (PCA) menggunakan PLINK v1.9 dan memvisualisasikan hasilnya melalui ggplot2. Hubungan filogenetik digambarkan menggunakan MEGA-CC [ 31 ] dengan 1000 replikasi bootstrap menggunakan metode neighbor-joining. Pohon filogenetik yang dihasilkan divisualisasikan dan diberi anotasi menggunakan server daring iTOL ( https://itol.embl.de/ ). Analisis komponen leluhur dilakukan menggunakan ADMIXTURE v1.3 [ 32 ].
Untuk mengevaluasi efektivitas chip ayam Shennong 1 40K dalam analisis sinyal seleksi, kami melakukan analisis berikut. Pertama, kami menggunakan pola diferensiasi genetik ( F ST ) [ 33 ] dan metode statistik berbasis haplotipe lintas populasi (XP-nSL) [ 34 ] untuk bersama-sama mengeksplorasi gen adaptasi dataran tinggi pada ayam Tibet dan gen produksi dan pertumbuhan telur tinggi pada ayam Rhode Island Red, dengan lima ayam lainnya (ayam dataran rendah, LAC) yang berfungsi sebagai populasi referensi. Dengan menggunakan pendekatan jendela geser (ukuran jendela 100 Kb dan langkah 30 Kb), kami mengidentifikasi 5% teratas wilayah genom yang diseleksi. Anotasi gen dilakukan menggunakan bedtools [ 35 ] berdasarkan GRCg6a 105 ( https://ftp.ensembl.org/pub/release-105/gtf/gallus_gallus/ ), diikuti oleh analisis pengayaan KEGG dan GO menggunakan database KOBAS ( http://kobas.cbi.pku.edu.cn/kobas3/ ).
3 Hasil
3.1 Desain Keripik Ayam Shennong 1 40K
Pertama, kami memilih kelompok pertama situs SNP dari WGS yang berisi 12.381.606 SNP dari ayam lokal Tiongkok. Dengan menggunakan jendela 30 Kb dan langkah 25 Kb, kami memberi skor pada setiap SNP dalam jendela tersebut, dengan hanya menyimpan SNP dengan skor tertinggi di setiap jendela. Setelah mengevaluasi situs SNP dengan alat perancang probe GenoBaits, kami memperoleh satu set 35.100 SNP sebagai kelompok pertama situs target.
Kelompok kedua dari situs target adalah untuk pan-genom. Kami memanfaatkan urutan pan-genom, seperti yang diuraikan dalam pekerjaan kami sebelumnya [ 17 ]. Kami memilih situs target berdasarkan daerah homolog yang mengapit lokus dan konten CG (CG%). Akibatnya, kami mengidentifikasi 5074 SNP. Kelompok ketiga adalah untuk penelitian GWAS sebelumnya; kami mengekstraksi 7419 situs GWAS dari Chicken QTL SNPdb, berhasil memetakan ulang 7100 situs ini ke genom referensi GRCg6a. Pasca evaluasi menggunakan perangkat lunak perancang probe GBTS, 4660 situs target GWAS memenuhi kriteria kami dan dimasukkan sebagai kelompok ketiga situs target. Dalam memilih kelompok keempat situs fungsional gen, kami berusaha untuk menyusun daftar lengkap mutasi kausal yang dilaporkan sebelumnya. Upaya ini menghasilkan 56 situs, yang mencakup pertumbuhan, berat, warna bulu, warna betis, warna daging, warna kulit, warna kulit telur, dan faktor-faktor lain yang dapat memengaruhi ekonomi produksi. Pemetaan ulang selanjutnya dari semua 56 situs ke genom referensi GRCg6a dan mengevaluasinya dengan alat desain probe, rangkaian kami disempurnakan menjadi 48 lokus fungsional (33 SNP dan 15 PAV) yang sesuai untuk penangkapan probe yang ditargetkan.
Akhirnya, kami menggabungkan dan menduplikasi empat kelompok situs target untuk menyusun situs target 40K ayam Shennong 1 (lihat Tabel 2 ). Situs target chip digambarkan pada Gambar 3A , sedangkan Gambar 3B secara grafis menggambarkan korelasi antara jumlah situs target dan ukuran kromosom.
| Kategori situs chip | Jumlah lokasi target di Shennong 1 ayam 40K |
|---|---|
| WGS | 35.100 orang |
| Pan-genom | 5074 |
| GWAS | 4660 |
| Situs fungsional gen (SNP dan PAV) | 48 |
| Total | 44.849 |

3.2 Perbandingan Keripik Shennong dan Keripik Lainnya dalam Hal Efektivitasnya terhadap Populasi Ayam Lokal
Untuk mengevaluasi kesesuaian chip ayam Shennong 1 40K untuk ayam kampung Cina, kami membandingkan lokus polimorfiknya dengan tiga chip genotipe ayam komersial yang banyak digunakan (Affymetrix, Illumina, dan Jingxin) di seluruh populasi ayam kampung Cina.
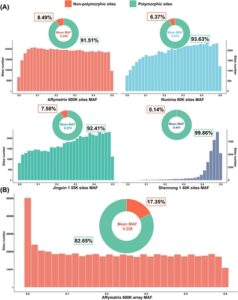
Hasil terperinci disajikan dalam Gambar 4 , di mana Gambar 4A dengan jelas menunjukkan bahwa chip Shennong 1 memiliki proporsi situs nonpolimorfik yang lebih rendah dibandingkan dengan tiga chip genotipe lainnya. Secara khusus, chip Affymetrix 600K memiliki hampir 10% situs nonpolimorfik, sedangkan Illumina dan Jingxin memiliki proporsi serupa yang berkisar antara 6% dan 7%. Lebih jauh lagi, untuk lebih memvalidasi temuan ini, kami menganalisis data genotipe array Affymetrix 600K yang tersedia untuk umum untuk ayam asli Tiongkok. Menurut statistik dari kumpulan data ini, 17,35% situs pada chip tersebut nonpolimorfik (Gambar 4B ). Secara keseluruhan, hasil ini menunjukkan bahwa lokus pada chip ayam Shennong 1 40K memiliki kemampuan beradaptasi yang baik untuk populasi ayam asli Tiongkok.
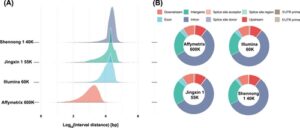
Mengenai keseragaman distribusi lokus, chip Shennong 1 chicken 40K juga menunjukkan kinerja yang unggul. Gambar 5A menunjukkan distribusi situs yang berdekatan untuk keempat chip. Dapat diamati bahwa chip Shennong 1 chicken 40K memiliki distribusi yang lebih terkonsentrasi dan simetris, sedangkan chip Illumina dan Jingxin memiliki distribusi yang sama, dan susunan Affymetrix lebih tersebar. Dalam hal anotasi fitur genomik (Gambar 5B ), hasil dari keempat chip relatif konsisten, dengan intron yang menyumbang proporsi tertinggi, diikuti oleh daerah intergenik, dan UTR menjadi yang terendah.
3.3 Kinerja Genotyping Ayam Shennong 1 40K Chip
Efisiensi chip dievaluasi dengan membuat genotipe sekelompok 204 ayam, yang mencakup 181 ayam lokal Cina dari enam ras dan 23 ayam Rhode Island Red.
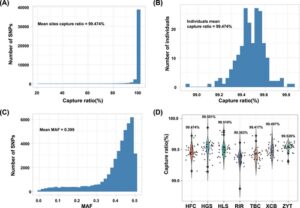
Pertama, kami menghitung tingkat penangkapan dari situs target, seperti yang ditunjukkan pada Gambar 6A . Tingkat penangkapan rata-rata adalah 99,474%. Khususnya, sebagian kecil situs menunjukkan rasio penangkapan di bawah 50%, terutama dalam situs pan-genom. Pada Gambar 6B , kami lebih lanjut memeriksa rasio penangkapan sampel untuk mengevaluasi efisiensi chip dalam menangkap data pada tingkat individu. Tingkat penangkapan sampel berkisar dari 98,891% hingga 99,831%, rata-rata 99,474%. Deteksi polimorfisme SNP oleh chip itu luar biasa (lihat Gambar 6C ), dengan MAF rata-rata 0,399. Dari semua situs, 44.187 SNP memiliki MAF > 0,05, yang mewakili 98,557% dari total. Lebih jauh, kami melakukan analisis tingkat penangkapan khusus ras untuk mengeksplorasi kemampuan deteksi chip pada berbagai ras ayam. Tingkat penangkapan rata-rata untuk setiap ras disajikan pada Gambar 6D . Di antara ras-ras tersebut, ayam Zhengyang tiga-kuning (ZYT) menunjukkan tingkat penangkapan tertinggi (99,526%), diikuti oleh Lushi (HLS: 99,510%), Gushi (HGS: 99,501%), Xichuan tulang hitam (XCB: 99,497%), Henan fighting (HFC: 99,474%), Lasa putih (TBC: 99,417%), dan Rhode Island Red (RIR: 99,363%), dengan semua ras melebihi rasio penangkapan 99%.
3.4 Pemanfaatan dalam Analisis Keanekaragaman Genetik dan Struktur Genetik
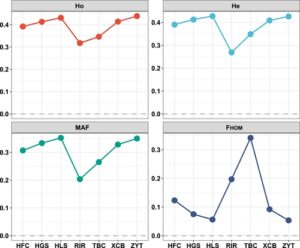
Seperti yang ditunjukkan pada Gambar 7 , kami menghitung empat parameter genetik, termasuk He, Ho, MAF, dan F HOM , untuk mengukur keragaman genetik tujuh ras ayam. Hasil perhitungan mengungkapkan konsistensi umum dalam tren Ho dan He di antara ras-ras, dengan RIR memiliki Ho terendah. Nilai Ho memberi peringkat ras-ras sebagai berikut: ZYT (0,439), HLS (0,431), XCB (0,414), HGS (0,413), HFC (0,392), TBC (0,346), dan RIR (0,318). He memberi peringkat mereka sebagai berikut: HLS (0,429), ZYT (0,426), HGS (0,413), XCB (0,409), HFC (0,391), TBC (0,349), dan RIR (0,269). Dengan demikian, tren MAF di antara ras-ras ini mencerminkan hasil heterozigositas, dengan MAF berkisar antara 0,204 dan 0,352. Ras-ras tersebut, yang diperingkat berdasarkan MAF, adalah sebagai berikut: HLS (0,352), ZYT (0,350), HGS (0,333), XCB (0,328), HFC (0,307), TBC (0,265), dan RIR (0,204). Koefisien perkawinan sedarah (F HOM ) menunjukkan korelasi negatif dengan parameter sebelumnya (Ho, He, MAF), yang menyoroti TBC sebagai ras yang memiliki tingkat perkawinan sedarah tertinggi (0,341), diikuti oleh RIR (0,197), HFC (0,123), XCB (0,0922), HGS (0,0750), HLS (0,0563), dan ZYT (0,0535).
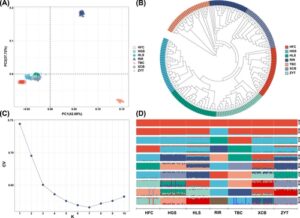
Dalam PCA (lihat Gambar 8A ), individu-individu dari ras yang sama khususnya mengelompok bersama. Ras yang dianalisis dibagi menjadi tiga kelompok: LAC (ayam dataran rendah: HGS, HFC, HLS, TBC, XCB), TBC, dan ayam RIR. Komponen utama pertama (PC1) membedakan LAC dari TBC dan RIR, sedangkan komponen utama kedua (PC2) memisahkan ayam TBC dan RIR. Demikian pula, analisis pohon filogenetik (Gambar 8B ) mengungkapkan pola pengelompokan yang kohesif, dengan individu-individu dari ras yang sama mengelompok bersama dalam cabang-cabang yang berbeda. Lima ras dataran rendah mengelompok menjadi cabang besar, yang secara jelas dipisahkan dari ayam TBC dan RIR. Dalam analisis campuran, K = 7 dibuktikan dengan kesalahan validasi silang (CV) terendah, yang konsisten dengan pengelompokan ras yang sebenarnya (Gambar 8C ). Divergensi evolusioner terbukti sebagai berikut: pada K = 2, RIR pertama kali diisolasi; pada K = 3, TBC; pada K = 4, HFC; pada K = 5, HGS; pada K = 6, ZYT; pada K = 7, XCB dan HLS diisolasi secara individual. Selain itu, dari K = 2 hingga K = 10, aliran gen diamati di antara lima ayam dataran rendah, dan kami juga mengamati peristiwa introgresi gen dari ayam RIR ke LAC (Gambar 8D ).
3.5 Pemanfaatan dalam Analisis Sinyal Seleksi
Kami pertama kali melakukan analisis F ST untuk mendeteksi sinyal seleksi diversifikasi antara TBC dan LAC (Gambar 9A ). Kami memilih 5% wilayah gen teratas berdasarkan F ST sebagai wilayah kandidat untuk seleksi. Selanjutnya, kami menggunakan XP-nSL untuk mengidentifikasi sinyal seleksi positif TBC (Gambar 9B ), mengambil 5% wilayah lainnya sebagai wilayah seleksi positif TBC. Setelah anotasi gen, kami memperoleh total 571 gen kandidat untuk seleksi positif TBC (lihat Tabel S2 ).
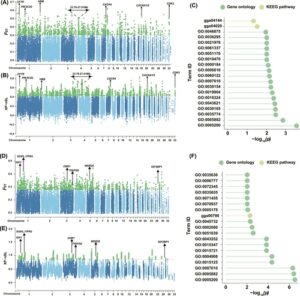
Kami selanjutnya melakukan analisis pengayaan fungsional KEGG dan GO pada gen kandidat. Gambar 9C menampilkan 20 entri teratas yang diperkaya secara signifikan. Semua entri yang diperkaya secara signifikan dapat ditemukan di Tabel S2 . Jalur KEGG yang diperkaya mencakup jalur pensinyalan kalsium (gga04020) dan endositosis (gga04144). Mengenai analisis pengayaan GO, terutama berfokus pada istilah GO seperti respons seluler terhadap peningkatan kadar oksigen (GO:0036295), transpor ion kalsium (GO:0006816), konduksi jantung (GO:0061337), konstituen struktural sitoskeleton (GO:0005200), dan regulasi positif sekresi insulin yang terlibat dalam respons seluler terhadap stimulus glukosa (GO:0000184). Ada enam gen ( CFTR, PIK3C2G, HBB, CXCR4, CACNA1G, dan CDK2 ) yang dikaitkan dengan adaptasi ketinggian tinggi atau fitur ras, dan mereka memiliki fungsi biologis yang masuk akal. Di antara mereka, CACNA1G dan CXCR4 diperkaya dalam jalur pensinyalan kalsium. Selain itu, dalam wilayah genomik yang membentang dari 23,76 hingga 28,91 Mb pada kromosom 4, sinyal diferensiasi dan seleksi yang berbeda ( F ST dan XP-nSL > 1% teratas) diamati. Gen pengkode protein yang dijelaskan dalam wilayah ini meliputi KLHL2, MSMO1, CPE, TLL1, SPOCK3, ANXA10, LOC422426, DDX60, PCDH18, dan SLC7A11 .
Demikian pula, kami juga melakukan analisis F ST pada ayam RIR dan LAC untuk mendeteksi sinyal seleksi yang beragam di antara keduanya (Gambar 9D ). Selanjutnya, kami menggunakan XP-nSL untuk mengidentifikasi sinyal seleksi positif RIR, dengan mengambil 5% daerah lainnya sebagai daerah seleksi positif RIR (Gambar 9E ). Setelah anotasi gen, kami memperoleh total 598 gen kandidat untuk seleksi positif RIR (Tabel S2 ).
Kami melakukan analisis pengayaan fungsional KEGG dan GO pada gen kandidat yang dipilih secara positif pada ayam RIR, yang menyajikan 20 entri teratas yang diperkaya secara signifikan (lihat Gambar 9F ). Dalam jalur KEGG, hanya biosintesis folat yang menunjukkan pengayaan signifikan (gga00790). Sedangkan untuk analisis pengayaan GO, terutama difokuskan pada jalur pensinyalan seperti membran akrosom (GO:0002080), aktivitas saluran pelepasan kalsium yang sensitif terhadap NAADP (GO:0072345), transpor anion organik yang tidak bergantung pada natrium (GO:0043252), aktivitas transporter transmembran anion organik yang tidak bergantung pada natrium (GO:0015347), aktivitas hormon (GO:0005179), dan lainnya. Semua entri yang diperkaya secara signifikan dapat ditemukan di Tabel S2 . Ada tujuh gen ( IGF1, SOX5, ITPR2, CNR1, TFAP2D, WDR25, dan IGF2BP1 ) yang diduga berhubungan dengan regulasi produksi telur.
3.6 Menggunakan Situs Fungsional untuk Evaluasi Breed yang Tepat
Untuk memulai, kami menggunakan dua situs fungsional (ukuran tubuh dan bulu bergaris) sebagai contoh untuk menunjukkan kemampuan chip untuk evaluasi ras yang akurat. Lokus ukuran tubuh adalah delesi segmen besar 3,2K, dan berdasarkan studi pan-genom kami sebelumnya, individu mutan (ALT) pada lokus ini menunjukkan laju pertumbuhan yang lebih tinggi daripada individu tipe liar (REF). Situs bulu bergaris sangat terkait dengan pola bulu bergaris pada ayam asli Tiongkok, dengan individu ALT menampilkan fenotipe bergaris. Gambar 10 menggambarkan frekuensi gen berdasarkan kedua lokus ini. Pada Gambar 10A , dapat diamati bahwa frekuensi alel ALT dari RIR dan HFC lebih tinggi daripada ras lain, sedangkan TBC, XCB, dan HLS memiliki frekuensi alel bulu bergaris yang lebih tinggi. ZYT, ayam berbadan kecil, memiliki frekuensi alel ALT yang relatif rendah di kedua situs. Gambar 10B mengilustrasikan distribusi frekuensi alel terperinci di kedua lokasi ini dan foto-foto ras ayam yang sesuai. Untuk data frekuensi alel terperinci dari setiap lokasi fungsional, silakan lihat Tabel S3 .

4 Diskusi
Bahasa Indonesia: Mengingat perkembangan terkini dalam teknologi genotyping, penggunaan penanda SNP genom-lebar untuk penilaian sumber daya genetik telah diterapkan secara luas dalam industri peternakan, khususnya dalam sapi [ 36 ], babi [ 37 ], bebek [ 38 ], dan ayam [ 28 , 39 ]. Breed asli cenderung menunjukkan sifat-sifat yang berbeda, seperti ketahanan penyakit yang sangat baik [ 40 ], menghasilkan produk daging dan telur yang unggul [ 41 ], dan menunjukkan ketahanan lingkungan yang lebih besar [ 42 ]. Akibatnya, penilaian genetik dan pelestarian breed asli ini memainkan peran penting dalam memastikan keberlanjutan sektor peternakan. Dalam studi ini, kami, untuk pertama kalinya, telah merancang chip genom cair spesifik (bernama chip Shennong 1 chicken 40K) untuk breed ayam asli Tiongkok. Chip tersebut menggunakan teknologi GBTS [ 12 ], yang merupakan strategi baru yang menggabungkan hibridisasi sekuens target dan teknologi sekuensing generasi berikutnya (NGS). Chip tersebut terdiri dari total 44.849 lokasi target, yang diperoleh dari empat sumber berbeda: data WGS, data pan-genom [ 17 ], data GWAS [ 24 ], dan data fungsional yang dilaporkan sebelumnya untuk sifat-sifat ekonomi. Untuk mengevaluasi secara sistematis penerapan chip Shennong 1 chicken 40K yang lebih unggul untuk ras ayam asli Tiongkok, kami melakukan analisis komparatif dengan tiga susunan genotipe ayam lain yang banyak digunakan dalam penelitian ini. Hasil akhir menunjukkan bahwa chip Shennong 1 chicken 40K menunjukkan kesesuaian yang lebih baik untuk ayam asli, dengan distribusi lokasi yang lebih seragam.
Selanjutnya, kami menilai kinerja genotipe chip menggunakan 204 individu dari tujuh ras ayam. Hasilnya menunjukkan bahwa chip mencapai rasio penangkapan rata-rata lebih dari 99% untuk situs target. Namun, ada rasio penangkapan yang relatif lebih rendah untuk situs spesifik pada kromosom W dan pan-genom. Kromosom W tidak ada pada individu jantan, sehingga menyebabkan tidak adanya situs target pada kromosom tersebut. Urutan pan-genom merujuk pada urutan spesifik yang tidak ada dalam genom referensi [ 17 , 43 ]. Demikian pula, laporan sebelumnya [ 7 ] telah menunjukkan rasio penangkapan rata-rata sekitar 29,2% untuk situs target pan-genom pada ayam, yang konsisten dengan hasil yang diperoleh dalam penelitian ini. Situs target chip memiliki frekuensi alel minor (MAF) rata-rata 0,399. Lebih jauh lagi, 98,557% situs memiliki MAF yang melebihi 0,05, yang menunjukkan tingkat universalitas yang tinggi di seluruh ras. Dalam analisis rasio penangkapan chip untuk berbagai ras, ditemukan bahwa bahkan untuk ras ayam Amerika (ayam RIR), rasio penangkapan chip masih mencapai 99,363%. Ini menunjukkan bahwa chip memiliki potensi aplikasi yang besar dan dapat digunakan untuk sebagian besar ras ayam, bukan hanya ayam lokal Cina.
Dalam analisis keragaman genetik, hasil dari empat parameter keragaman genetik, Ho, He, MAF, dan F HOM , relatif konsisten di antara tujuh ras. Perlu dicatat bahwa nilai Ho, He, dan MAF tampaknya secara umum lebih tinggi dibandingkan dengan yang dihitung berdasarkan WGS. Hal ini dapat dikaitkan dengan pemilihan preferensial situs target chip dengan MAF yang lebih tinggi. Oleh karena itu, pengamatan ini sejalan dengan harapan, dan pada dasarnya, tidak berarti untuk secara langsung membandingkan nilai yang diperoleh dari set penanda SNP yang berbeda (seperti data resequencing genom dan data chip). Dalam analisis struktur populasi, chip berhasil mengelompokkan ras, dan hasil dari PCA, pohon filogenetik, dan analisis campuran konsisten, yang menunjukkan kekokohan situs target. Menariknya, dalam analisis campuran, kami mengamati aliran gen antara ayam asli Cina dan ayam RIR. Peristiwa aliran gen serupa juga telah diamati antara babi asli Cina dan ras babi asing [ 44 ]. Aliran gen sering kali memberikan kontribusi terhadap evolusi spesies dan kemampuan mereka untuk beradaptasi dengan lingkungan yang berbeda [ 45 ]. Hal ini menunjukkan bahwa ayam RIR mungkin telah mentransfer gen tertentu, seperti yang terkait dengan produksi atau pertumbuhan telur, ke ayam asli Tiongkok.
Dalam analisis sinyal seleksi, kami menggabungkan F ST dan XP-nSL untuk menyelidiki sinyal seleksi positif pada ayam TBC dan RIR. Pada TBC, kami mengidentifikasi 571 gen kandidat, di antaranya enam gen ( CFTR, PIK3C2G, HBB, CXCR4, CACNA1G, dan CDK2 ) sebelumnya telah dilaporkan sebagai gen adaptif dataran tinggi. CFTR terkait dengan adaptasi paru-paru yak dan Tibet terhadap hipoksia dataran tinggi [ 46 , 47 ]; PIK3C2G terkait dengan respons hipoksia [ 48 ], dan secara signifikan diperkaya dalam pensinyalan yang dimediasi fosfatidilinositol (GO:0048015). PIK3C2G telah diidentifikasi sebagai gen adaptif dataran tinggi pada babi hutan Tibet dan domba Tibet [ 48 ]; HBB telah diidentifikasi sebagai gen kandidat untuk adaptasi hipoksia pada Tibet dan mastiff Tibet [ 49 ]; CXCR4 [ 50 ], dan CACNA1G [ 51 ] telah dilaporkan dalam penelitian sebelumnya tentang adaptasi dataran tinggi pada ayam Tibet sebagai gen yang terkait dengan respons hipoksia, dan gen tersebut secara signifikan diperkaya dalam jalur pensinyalan kalsium. Penelitian sebelumnya tentang adaptasi dataran tinggi pada ayam Tibet telah melaporkan bahwa gen tersebut terlibat dalam jalur pensinyalan kalsium [ 50 , 51 ]. Oleh karena itu, CXCR4 dan CACNA1G dapat dianggap sangat kredibel sebagai gen kandidat untuk adaptasi dataran tinggi pada ayam Tibet. CDK2 adalah gen adaptif untuk adaptasi dataran tinggi pada domba Tibet [ 52 ] dan mastiff Tibet [ 53 ], yang berpartisipasi dalam apoptosis miosit jantung yang diinduksi hipoksia. Lebih jauh, kita dapat dengan jelas mengamati sinyal seleksi ( F ST dan XP-nSL > top 1%) pada kromosom 4 (23,76–28,91 Mb), dan melalui anotasi gen, kita telah mengidentifikasi 10 gen pengkode protein. PCDH18 menunjukkan korelasi kuat dengan radiasi matahari musim panas [ 54 ]. SLC7A11 dikaitkan dengan pembentukan warna bulu coklat-hitam dan dapat berkontribusi terhadap adaptasi ayam Tibet terhadap tingkat radiasi ultraviolet yang tinggi di lingkungan dataran tinggi [ 55 , 56 ].
Dalam sejarah, RIR dibiakkan sebagai ayam dwiguna untuk produksi daging dan telur [ 57 ]. Saat ini, mereka terutama digunakan sebagai induk untuk ayam petelur komersial. Oleh karena itu, RIR menunjukkan kinerja yang jauh lebih baik dalam hal produksi telur dan pertumbuhan dibandingkan dengan ras ayam asli Cina. Kami mengidentifikasi 598 gen kandidat, di antaranya tujuh gen ( IGF1, SOX5, ITPR2, CNR1, TFAP2D, WDR25, dan IGF2BP1 ) sebelumnya telah dilaporkan sebagai gen produksi telur dan pertumbuhan yang tinggi. Dalam penelitian sebelumnya, SOX5 [ 58 ], IGF1 [ 58 ], dan IGF2BP1 [ 17 ] telah diidentifikasi sebagai gen kunci yang terlibat dalam pertumbuhan. Beberapa penelitian juga menemukan korelasi antara tingkat ekspresi gen IGF1 dan sifat bertelur pada ayam [ 59 ]. Gen IGF2BP1 mungkin telah memainkan peran penting dalam pengembangan garis ayam tipe telur [ 60 ]; Gen SOX5 dan CNR1 telah dikonfirmasi sebagai pengatur produksi telur pada ayam Rhode Island Red dalam studi RNA-seq kami sebelumnya [ 61 ]; ITPR2 [ 62 ], TFAP2D [ 63 ], dan WDR25 [ 64 ] telah diidentifikasi sebagai gen kandidat untuk kualitas telur dan produksi telur, masing-masing, pada ayam melalui GWAS.
Dengan mengintegrasikan situs fungsional yang dilaporkan sebelumnya [ 17 ] dan memanfaatkan chip untuk genotipe yang efisien dan berthroughput tinggi, evaluasi akurat ras dapat dilakukan dengan menganalisis frekuensi alel dari berbagai genotipe. Ini memberikan wawasan lebih jauh ke dalam produksi dan karakteristik ekonomi dari ras tertentu. Mengamati distribusi frekuensi gen dapat memandu pemulia dalam memutuskan apakah akan fokus pada pembiakan dalam populasi yang ada atau memperkenalkan garis keturunan baru. Kami menghitung frekuensi alel dari 48 situs fungsional dalam tujuh ras. Studi ini mengambil berat badan dan situs bulu bergaris sebagai contoh untuk mengeksplorasi sifat karakteristik dari tujuh ras ayam. Untuk situs yang berhubungan dengan ukuran tubuh, RIR dan HFC menunjukkan frekuensi alel ALT yang lebih tinggi dibandingkan dengan ras lain, yang menunjukkan ukuran tubuh mereka yang lebih besar. Untuk situs yang berhubungan dengan bulu bergaris, ras seperti TBC, XCB, dan HLS menunjukkan frekuensi alel ALT yang lebih tinggi, yang menunjukkan rentang warna bulu yang lebih beragam pada ras ini, yang dapat menyediakan sumber daya genetik yang berharga untuk mengembangkan garis keturunan baru.
5 Kesimpulan
Singkatnya, cakupan situs polimorfik dari chip ayam Shennong 1 40K mencapai 99,86% pada populasi ayam asli Tiongkok, yang lebih tinggi dari 6%–8% yang diamati pada chip gen yang tersedia secara komersial. Hasil ini menunjukkan kemampuan adaptasi dan ketahanannya yang luar biasa terhadap berbagai ras ayam asli Tiongkok yang beragam secara genetik. Lebih jauh, penggabungan data pan-genom dan tipe multivarian meningkatkan potensi aplikasi chip ini untuk populasi ayam non-Tiongkok. Hal ini akan mendorong evaluasi ilmiah dan tepat terhadap ayam asli Tiongkok, eksplorasi sumber daya genetik, serta upaya pembiakan dan konservasi.


Leave a Reply